Metabarcoding de DNA ambiental: un enfoque para el seguimiento de la
biodiversidad
Environmental DNA metabarcoding: an
approach for biodiversity monitoring
Estudios de eDNA
Cinthia
Yedith Padilla-García, Fátima Yedith Camacho-Sánchez, Miguel Ángel Reyes-López*
*Correspondencia:
mareyesl@gmail.com/ Fecha de recepción: 13 de diciembre de 2020/Fecha de
aceptación: 25 de junio de 2021/Fecha de publicación: 27 de julio de 2021.
Instituto
Politécnico Nacional, Centro de Biotecnología Genómica, Conservation
Medicine Laboratory, boulevard del maestro s/n
esquina Elías Piña, col. Narciso Mendoza, Reynosa, Tamaulipas México. C. P.
88710.
Resumen
El término ácido desoxirribonucleico ambiental o DNA ambiental (eDNA) se
acuñó para definir al ácido desoxirribonucleico (DNA) que se puede recuperar
o detectar del ambiente (por ejemplo: suelo, aire o agua) sin
necesidad de que el espécimen esté físicamente presente. El objetivo del presente trabajo fue analizar y ejemplificar los
usos, aplicaciones y potencial del eDNA. El estudio del eDNA es
utilizado para la evaluación de especies, que va desde la reconstrucción
histórica de sus comunidades, la restauración del ecosistema, hasta la salud humana, lo que lo convierte en una herramienta
versátil e importante para el futuro en investigación,
permitiendo estudios de conservación, taxonómicos o de reconstrucción filogenéticos.
Para lograr esto, se usa el procedimiento de metabarcoding,
el cual se basa en obtener DNA de cualquier origen (en este caso eDNA), en ausencia física o no del organismo, con apoyo de la reacción en cadena de la polimerasa (PCR), para finalmente, secuenciarlos
y obtener códigos de barras. Los estudios de eDNA
probablemente se constituirán como un enfoque esencial para diversas tareas científicas
no solo en el seguimiento de la biodiversidad, sino en el análisis de la salud
humana o la generación de códigos de barras de DNA.
Palabras clave: eDNA,
biodiversidad, PCR, secuenciación.
Abstract
Environmental deoxyribonucleic
acid or environmental DNA
(eDNA) is a term coined to define the deoxyribonucleic
acid (DNA) that can be
recovered or detected in an environment (for
example: soil, air, or water) sample,
but not specimen physically present
(metagenome). The objective of this review was to understand, analyze, and define the uses, applications,
and potential of eDNA. eDNA
brings together several fields ranging
from ecological assessment for historical community
reconstruction, ecosystem restoration to human health, making it
extremely versatile and important for the future in research, such as studies
of conservation, taxonomic, or phylogenetic
reconstruction. To achieve this, the
metabarcoding procedure is used, which is based on obtaining DNA of any origin
(in this case eDNA), in the physical absence or not of organisms, with the polymerase chain reaction (PCR), to finally sequence them and develop
barcodes. The application of eDNA in biological
research seems almost limitless, but
it requires collaboration and
coordination of scientific teams. eDNA
studies will probably become an essential
tool for different scientific tasks not only in monitoring biodiversity,
but also in human health analysis or
in the development of DNA barcodes.
Keywords: eDNA, biodiversity, PCR, sequencing.
INTRODUCCIÓN
El estudio y conocimiento de la diversidad genética, el cual
detecta las variaciones heredables que pudieran ocurrir en cada organismo,
entre los individuos de alguna población y entre poblaciones de una misma
especie, cobra relevancia desde hace varias
décadas por la diversidad de usos y trabajos que presenta. Para lograr
implementar ese tipo de propuestas se requiere
de una interrelación entre varias áreas, disciplinas o ciencias. Así, la
genética y la evolución se agrupan para
entender, describir y dar las pautas en trabajos de biodiversidad, para
que sea estudiada o entendida y pueda ser
protegida y conservada (Rimieri,
2017).
Alrededor del mundo se están perdiendo diversas especies (Scheffers y
col., 2012; Raven, 2020) de flora y fauna
silvestre debido a que el ser humano modifica los ambientes a través de la pérdida del hábitat, la contaminación ambiental
(presencia de componentes nocivos en el medio ambiente, que pueden ser
químicos, físicos o biológicos, ya sea en un entorno natural o artificial), la
sobreexplotación, la introducción de especies invasoras, el cambio climático,
entre otros (Koslowski y col., 2020). Además del
empleo de algunos métodos convencionales (como quemas, generación de dióxido de
carbono por industrias, incendios o combustión de automóviles, uso de
plásticos, y otros) que aceleraron las alteraciones del ambiente y tal vez la
pérdida de alguna especie, por lo que es necesario desarrollar metodologías más
precisas que en algunos casos logren distinguir dos especímenes o especies entre sí, para finalmente detectarlos,
diferenciarlos y contabilizarlos (Yildiz y col., 2019).
Aunque aún no existe la información completa de la magnitud
de dicha pérdida de la biodiversidad, hay un acuerdo internacional llamado Plan Estratégico para la Biodiversidad 2011-2020,
que marca los procedimientos para tratar de detener y revertir la pérdida de la
biodiversidad de acuerdo al Programa de Naciones
Unidas para el Medio Ambiente (UNEP, por sus siglas en inglés: United Nations Environment Programme) (UNEP,
2011). Estas acciones dependen en gran medida
del monitoreo biológico, que permite obtener datos de la especie, lo que
se logra normalmente visualizándolas y cuantificándolas individuo por individuo
basándose en diversos caracteres morfológicos. El dilema se presenta cuando las
especies son similares o idénticas morfológica o anatómicamente (Thomsen y Willerslev, 2015; Deiner y col., 2017). Los errores que se presentan son muy
comunes en la clasificación de algunos taxones debido a que se requiere personal
altamente entrenado, por lo que se implementan otras técnicas para su
identificación y futuro uso de la
información (Bortolus, 2008; Hernández-González
y col., 2016), como la caracterización molecular.
El campo de la biología molecular cobró relevancia desde el
año 2000, dado que entre sus aplicaciones está el estudio de los genomas de
organismos, desde un microorganismo hasta organismos superiores, como los
humanos. Para entender cómo el estudio genómico de los ácidos nucleicos es
utilizado en biodiversidad es necesario señalar que una molécula clave para
esos estudios es el ácido desoxirribonucleico (DNA, por sus siglas en inglés: deoxyribonucleic acid), la cual está
compuesta por átomos en forma de doble hélice y tiene cuatro bases
nitrogenadas llamados nucleótidos: adenina, guanina, citosina y timina. El DNA
se encarga de trasmitir la información
genética (Vázquez-Ramos, 2016; Koch y
col., 2020) y es útil para identificar individuos al comparar secuencias de origen distinto o no.
Con los estudios moleculares del DNA se puede complementar la
morfología o la anatomía de especímenes o
seres humanos, respectivamente, con lo se obtiene una mayor precisión de
la identificación de sujetos (Pizarro, 2003), o mediante el uso de ácido
ribonucleico (RNA, por sus siglas en inglés: ribonucleic
acid), con el
mismo principio de detección del DNA. Estos estudios requieren de cantidades pequeñas de muestra (micro o nanogramos) de ácido nucleico (DNA o RNA) usada para su
análisis o amplificación; el protocolo es rápido (menos de 3 h) y está basado
en la reacción en cadena de la polimerasa (PCR, por sus siglas en inglés: polymerase chain reaction) (Zhu y col., 2020), o de un par de días para otras
técnicas más elaboradas, comparadas con los cultivos de bacterias u hongos (de 1 d
a 4 d o más días para hongos de lento
crecimiento) (Austin, 2017); versátil (múltiple y/o diferentes tipos de
muestras residuales) y, en algunos casos, barato (cuando se consideran pruebas
masivas) (Deiner y col., 2017). Asimismo, es utilizada es la secuenciación (Mani,
2020), la cual cuenta con variantes, desde secuenciación química,
enzimática, de electroforesis capilar, como la secuenciación tradicional, que
por muchos años fue la más común, llamada secuenciación de Sanger,
enzimática, capilar o de primera generación (Sanger y
col., 1977; van-Dijk y col., 2018), que consume mucho
tiempo, y se puede analizar especímen por especimen para obtener una secuencia a la vez. Ésta se basa
en la terminación de la amplificación o extención de cadenas de ácidos nucleicos, lo cual produce cadenas de
diferentes tamaños que se pueden agrupar por
el nucleótido común en donde se terminó la reacción enzimática. Los
avances en secuenciación pasaron de secuenciar unas cuantas muestras o
fragmentos, hasta análisis de miles o millones de fragmentos en un solo ensayo por medio de secuenciación
de siguiente generación (NGS, por sus siglas en inglés: next
generation sequencing), por ejemplo. Esta técnica revolucionó las
ciencias biológicas debido a su rendimiento masivo, escalabilidad y velocidad,
lo que permite investigaciones en una amplia variedad de aplicaciones y
estudios en sistemas biológicos a un nivel
nunca antes posible (van-Dijk y col., 2018).
Por un lado, se tienen las secuencias de
las especies o especímenes que se quiere identificar o estudiar
y, por el otro, se debe saber si fueron o no identificados antes y si dicha
secuencia ya está reportada. Con la información anterior, ambas secuencias son
comparadas o alineadas. En el caso de que ya estén publicadas las secuencias se
puede recurrir a sitios en la web que cuentan con herramientas bioinformáticas,
como el GenBank (Benson y
col., 2013), o mediante el uso de programas bioinformáticos
(ambas maneras se describen posteriormente).
Así, los análisis genómicos se basan en metodologías
moleculares, las cuales muestran avances importantes en las técnicas de
identificación de individuos o especímenes, lo que permiten detectarlos incluso
sin tener evidencia física o visual del organismo (Andruszkiewicz
y col., 2017b). Cabe señalar que estos estudios pueden dividirse en análisis
con o sin amplificación de los ácidos nucleicos, llamados estudios de metabarcoding
y metagenómicos, respectivamente.
En paralelo, las características de especies de vida
silvestre u organismos que se cree extintos, escasos, escurridizos a simple
vista o que no se pueden cultivar por métodos tradicionales, pueden inferirse
mediante la detección de su DNA. A partir de este punto, el concepto de DNA ambiental (eDNA, por sus siglas en inglés: environmental
DNA) emergió, describiéndose como el DNA que
se puede obtener de muestras tomadas en el ambiente sin estar presente
algún individuo físicamente (Taberlet y col., 2012; Ruppert y col., 2019), es decir, a partir de muestras de
piel, pelo, fluidos u otros tejidos o materiales, como el suelo, aire o agua.
Por lo que detectar el DNA y conocer a qué organismo
o entidad biológica se parece es importante para obtener información
sobre especies, poblaciones y comunidades (Thomsen y Willerslev, 2015) habitantes en un área específica o en
determinado momento del tiempo.
Asegurar el éxito de la detección del eDNA
dependerá de saber manejar la muestra recolectada y el posterior aislamiento e
integridad del material genético obtenido de
dicha muestra del medio ambiente (Andruszkiewicz
y col., 2017a), hasta su identificación.
Este trabajo tuvo como objetivo dar a conocer el estado
actual, usos y aplicaciones del estudio del eDNA, sus bondades, características, dificultades y
aplicaciones en el estudio de la biodiversidad, salud, ambiente y de muestras
antiguas.
I. eDNA
El término eDNA fue introducido en
1980, utilizado para detectar y describir comunidades de microorganismos en
sedimentos marinos (Ivanova y col., 2019). En 1990 fue clasificado de acuerdo
al tamaño de la partícula: DNA mayor a 0.2 m fue descrito como DNA particulado, y menor a esto, DNA disuelto (Díaz-Ferguson y Moyer, 2014).
Para profundizar en el concepto: el DNA está contenido en
materiales liberados por los organismos hacia el ambiente, los cuales pueden
ser desde heces, mucosidades, células de la
piel, organelos, gametos, inclusive, hasta DNA
extracelular (Deiner y col., 2017). Como ya se
demostró en diversos estudios, la técnica puede proporcionar información de macroorganismos extintos u organismos que hace tiempo transitaron
por un punto dado pero que ya no están en el área. Esto se debe a que los materiales
orgánicos conservan cadenas cortas de DNA que se mantienen en el ambiente por largos espacios de tiempo (Ficetola y
col., 2008).
Sin embargo, la obtención de este tipo de DNA no es fácil, ya
que hay factores ambientales que pudieran
afectarlo (Hänfling y col., 2016), como la
temperatura, el pH, la conductividad, o comunidades microbianas capaces
de degradar el DNA (Andruszkiewicz y col., 2017a).
Además, dependiendo del tipo de la muestra, como las heces, agua o sedimentos orgánicos, las concentraciones obtenidas
en la extracción de DNA variarán, lo cual dependerá del espécimen, de su dieta,
la estación del año, la densidad, que se encuentren en el agua o en el suelo,
el tipo de sedimento (Valdez-Moreno y col., 2019), incluso de su pelaje. Los rangos de concentración de DNA que se
puede obtener varían de 1 ng/L a 10 ng/L son suficientes
para realizar un PCR de la muestra.
II. Bases técnicas del uso del eDNA
Para el estudio y análisis de eDNA
se pueden seguir protocolos previamente
publicados. En la metodología de Díaz-Ferguson
y Moyer (2014) se argumenta que para la detección del
eDNA se requiere el desarrollo de marcadores genéticos
específicos para cada objetivo del trabajo.
Los marcadores genéticos son secuencias conocidas dentro del ácido nucleico a estudiar y de las cuales se conoce
normalmente su posición. Una vez detectados esos marcadores, se sintetizan sondas moleculares complementarias
a ellos, para posteriormente ser empleados
en diversos métodos, como el PCR, el
cual amplifica millones de veces el fragmento
de interés a estudiar, para enseguida ser visualizado a través de una electroforesis en geles de
agarosa o poliacrilamida (Díaz-Ferguson y Moyer, 2014; Salipante y
Jerome, 2020).
En algunas otras metodologías, como la que señalan
Valdez-Moreno y col. (2019) y que se resume
en la Figura 1, se utiliza el término metabarcoding, que
implica la recolección de muestras ambientales (presencia física o no de los especímenes a estudiar), la extracción de DNA, amplificación
por PCR para un gen de interés, por ejemplo, el gen mitocondrial de la citocromo
oxidasa-I (COI por sus siglas en inglés:
cytochrome c oxidase I) y secuenciación; también se
puede emplear el procedimiento de “metage-nómica”,
para la cual no requiere de la visualización física del espécimen del medio
ambiente y que, además, implica una colección de genes amplificados y
secuenciados (pero sin pasar por un PCR, a
diferencia del metabarcoding, que sí lo
requiere). Es decir, mientras la metagenómica analiza
secuencias nucleares, cloro-plastídicas o
mitocondriales diversas sin el uso previo de PCR para secuenciar, el metabarcoding se basa en el mismo principio pero se apoya del PCR y utiliza secuencias conservadas, universales y con regiones variables, que pueden
servir como código de barras.
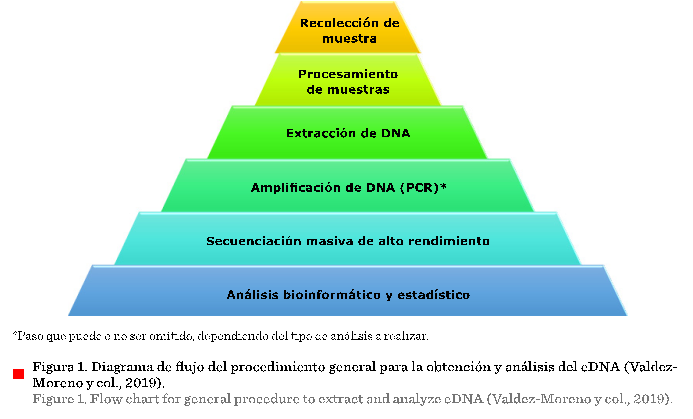
Con lo anterior, se puede caracterizar la biodiversidad presente
en una región dada, logrando la detección de especies o especímenes mediante la
generación de sus secuencias específicas dentro del genoma o genoma mitocondrial
del organismo, que servirán como una etiqueta molecular para la identificación
de especies. Una serie de ejemplos de la versatilidad del estudio de eDNA se presenta como parte
de la amplitud de este tipo de estudios. A modo de muestra, en el mar costero
en Japón y en un río en Indiana, EE. UU., el DNA se aisló, se secuenció y se
consiguió determinar, a partir de los restos de especímenes, el origen de una
especie sin haber tenido conocimiento previo de su existencia en el lugar (Deiner y col., 2016; Lacoursière-Roussel
y col., 2016; Valentini y col., 2016; Cowart y col., 2018; Valdez-Moreno y col., 2019 ).
Otros ejemplos de los usos de eDNA
son en la eutrofización (el proceso de contaminación más importante de las
aguas en lagos, balsas, ríos, embalses, entre otros) de las cuerpos de agua,
tanto dulce como de mar, la cual es causada por los efectos de las actividades
humanas y los aportes de nutrientes, lo que podría
provocar la proliferación de algas nocivas (HAB, por sus siglas en
inglés: harmful algal blooms), disminución de
la calidad del agua, reducción de la biodiversidad, lo cual podría
amenazar la salud humana (Liu y col., 2020); con la técnica podría identificarse los organismos presentes, benéficos o no y sus implicaciones
en el ecosistema. Además, se podría estudiar el monitoreo de la biodiversidad, el eDNA podría
usarse para estudios genéticos de poblaciones, lo cual es un campo emergente
que ofrece perspectivas interesantes,
sin embargo, habrá que seguir desarrollándolo (Adams y col., 2019).
Para
obtener una muestra y su análisis como eDNA, a continuación se describen varios ejemplos donde se observa el
uso de muestras de diferentes fuentes y
orígenes, y su análisis.
II.1. Recolección de muestra de agua
Uno de los vehículos más comunes en el que se encuentra
muestras de DNA o material con potencial DNA es el agua. De acuerdo a estudios
realizados, donde se describe que la mayoría de los animales terrestres que se
acercan a cuerpos de agua disponibles para beberla,
tienen contacto de su saliva, tejido de su
hocico o pelo y que éstos pueden caer y encontrarse en dicho fluido, es importante describir el procedimiento
de cómo detectarlos. Por lo que se señala en
general, el procedimiento marca que las muestras se tomen de la zona
litoral a no más de 0.5 m, para posteriormente almacenarse en botellas estériles,
aunque la toma de la muestra se puede realizar directamente de agua dulce,
salobre, subterránea y de cuerpos tanto lénticos como lóticos.
Equipo y muestras deben ser manipulados utilizando guantes, para evitar contaminar
(material con DNA o RNA de las personas que manejan la muestra, de bacterias
del ambiente o de otras muestras cercanas) y
colocarlas en hielo, para su posterior procesamiento. Todo el equipo de
muestreo y filtrado se debe lavar con una solución de hipoclorito de sodio (llamada también lejía o cloro) comercial al 10
% antes de su uso. Las muestras de agua tienen que ser recolectadas en envases
de polietileno de aproximadamente 500 mL. En paralelo,
dos muestras testigos negativos (agua destilada)
deben ser colectadas en los recipientes, para hacer el seguimiento de
una potencial contaminación durante el muestreo y el transporte del agua (Ushio y col., 2017;
Valdez-Moreno y col., 2019). En México no se requieren permisos oficiales
para la colecta de este tipo de muestras, ya que son del ambiente y no de
vertebrados o de especies protegidas por las
leyes mexicanas, pero en caso de que se produzca una perturbación en el sitio de muestreo o que se utilicen,
aprovechen o descarguen aguas residuales sí se requeriría de un permiso oficial
de acuerdo al Diario Oficial de la Federación
(DOF, 2020) o lo procedente de acuerdo con las leyes y regulaciones del país o
región de que se trate.
II.2. Procesamiento de muestra de agua
Las muestras de agua se deberán filtrar dentro de las 7 h de colectadas,
para evitar que se degrade el material genético. Los artículos de filtrado más
utilizados son los filtros de fibra de vidrio y los de nitrato de celulosa, con
diferentes tamaños de poros. Los de poros más grandes permiten mayores
volúmenes de agua y los de poros más pequeños capturan más partículas, pero
limitan el volumen y la velocidad de filtración (Muha
y col., 2019). Por ejemplo, existen filtros circulares de membrana de nitrato
de celulosa Whatman, con tamaños de poros de 0.45 m y
diámetro 47 mm, y filtros de fibra de vidrio de borosilicato
Advantec GA55 con tamaño de poros de 0.6 m y diámetro de 47 mm.
Ya en el laboratorio o lugar donde se procesará la muestra, se debe trabajar en
un área estéril y tomar medidas para evitar la contaminación ambiental, por lo
que habrá que cambiarse los guantes entre cada muestra o manejo de muestra,
usar cubrebocas y, de ser posible, diferentes
cuartos, tanto para la extracción del DNA como para la amplificación. Al
terminar, el material filtrado se almacena a - 18 °C (Cowart
y col., 2018; Valdez-Moreno y col., 2019).
II.3. Extracción del DNA
Para la extracción del DNA se pueden
utilizar estuches comerciales (MoBio) normalmente vendidos como PowerSoil®
DNA Isolation Kit - Qiagen,
especiales para muestras de suelo, con indicaciones
del fabricante que deberán seguirse. Este método usa filtros, a
diferencia de otros que no los requieren.
El procedimiento no dura más de 10 min. Posteriormente se debe incubar a 4 ºC el DNA obtenido. Otros estuches comerciales (Muha y col., 2019) son DNA Qiagen
DNeasy Blood & Tissue (protocolo para manchas de sangre seca), usado para
múltiples muestras, como tejido o sangre total. Uno más es Nexttec (Nexttec Biotechnologie GmbH, Alemania),
con mayores ventajas debido a que evita una posible contaminación del DNA y que
es de un solo paso entre la digestión (ruptura de paredes celulares y proteínas
mediante enzimas o reactivos químicos) de la muestra y la elución final del
DNA. También se puede usar el NucleoSpin® Soil (Soliman y col., 2017), el
cual usa filtros y tiene una alta eficiencia en la obtención del DNA. Derivado
de este último estudio, se recomienda para la
estandarización el uso de diferentes individuos y por triplicado la
extracción de DNA, lo que podría ser una solución
para alcanzar un mejor protocolo para los estudios de metagenómica del suelo.
Finalmente, se pueden usar procedimientos tradicionales para la extracción de
DNA, como el de fenol: cloroformo (Green y Sambrook, 2017).
II.4. Amplificación del DNA
Para el estudio de las muestras de DNA existen diferentes
zonas blanco que habitualmente se usan como regiones (segmentos de DNA) de amplificación. Varios son los ejemplos: COI, para metazoos, y la cadena grande de bisfosfato de ribulosa carboxilasa (rcbL), para las plantas,
son los estándares establecidos por el código de barras de la vida.
Otras regiones usadas son el RNA ribosómico 12S o el 16S, que pueden ser más
apropiadas para diferentes taxones; para hongos se emplea ITS (por sus siglas en inglés: internal transcribed
spacer) y en bacterias el 16S. Para ciertos eucariotas
también se usa el 18S rRNA (Epp
y col., 2012; Taberlet y col., 2012; Gibson y col., 2015; Creer y col., 2016; Deiner
y col., 2017; Hering y col., 2018).
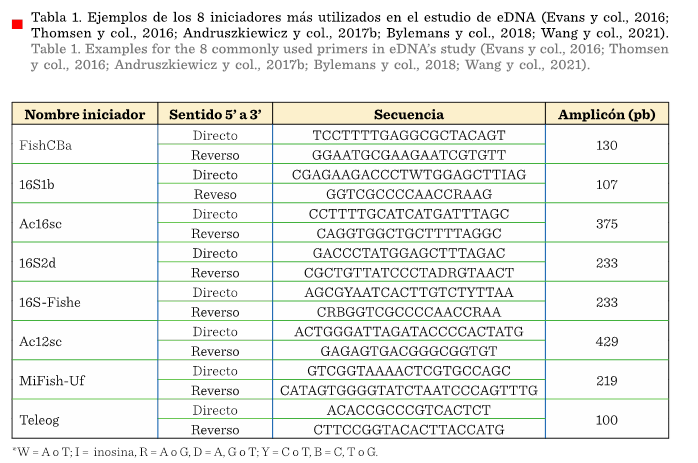
Los iniciadores
para el metabarcoding de eDNA (Tabla 1) deben
ser lo suficientemente cortos como para amplificar muestras degradadas,
idénticas pero variables entre especies y flanqueadas por regiones altamente
conservadas (segmentos de DNA que conservan su secuencia intacta a través del
tiempo) para amplificar una variedad de especies sin sacrificar la especificidad
del grupo objetivo. Se debe tomar en cuenta que se requieren procesos para asegurar
que el producto amplificado sea real o verdadero y no falsos negativos, por lo
que se realizan múltiples réplicas de PCR (realizar la reacción
múltiples veces) y de réplicas biológicas (colectar más de una vez las
mismas muestras biológicas).
Una metodología de amplificación que se usa con buenos
resultados es realizar un PCR de dos pasos. En el primero se utilizan iniciadores
convencionales y en el segundo, iniciadores de fusión con identificadores moleculares
únicos (UMI, por sus siglas en inglés: unique
molecular identifiers) o los códigos de barras moleculares
(MBC, por sus siglas en inglés: molecular barcodes),
que son secuencias de ácido nucleico cortas o “etiquetas” moleculares, útiles
en los protocolos de preparación de bibliotecas de secuenciación de siguiente
generación. Después de la primera ronda, los productos de PCR se diluyen, para
pasar a la segunda ronda de PCR. Finalmente, se purifica el DNA y se secuencia.
II.5. Análisis Bioinformático
Una vez obtenidas las secuencias, se comparan, resguardan y
analizan. El primer paso pudiera ser subir las secuencias a una biblioteca mundial
de secuencias. Existen diferentes bases de datos, una de las más conocidas es
la del código de barras de la vida (BOLD, por sus siglas en inglés: barcode of life bata system). En
ésta, cada secuencia cargada es clasificada y almacenada, para posteriormente
ser comparada con secuencias ya existentes en el sistema. Una utilidad de lo
anterior puede ser la clasificación de grupos de individuos relacionados, lo cual es conocido como unidad taxonómica operacional (OTU, por sus siglas en inglés: operational taxonomic unit), es decir, las
secuencias son agrupadas por similitud de secuencias de DNA de un gen marcador
taxonómico específico. Otra base de datos es la que se encuentra en el GenBank, que indica estar diseñada para proporcionar y
fomentar el acceso de la comunidad científica a la información de las secuencias
de DNA más actualizadas y completas, y forma
parte de lo que es el sitio web de El
Centro Nacional de Información Biotecnológica (NCBI, por sus siglas en inglés: National Center for Biotechnology
Information), que promueve la
ciencia y la salud al proporcionar acceso a información biomédica y genómica.
Ello ayudará a determinar si la secuencia problema secuenciada existe o no en
dicha base de datos, si es una secuencia conocida, o es la primera vez que se
detecta.
Por otro lado, derivado de la generación
de cientos, miles o millones
de secuencias por análisis, fueron creados sitios web para reunir tal
información, como es el caso de mBRAVE (multiplex barcode research and visualization environment), una plataforma multiusuario que admite el
almacenamiento, la validación, el análisis y la publicación de numerosos proyectos
basados en instrumentos de secuenciación de alto rendimiento (HTS, por sus
siglas en inglés:
high-throughput sequencing). Este sistema se basa en la plataforma BOLD para respaldar la identificación y el descubrimiento de especies para
los datos de HTS (Ratnasingham, 2019).
El procesamiento y la subida de los
datos a ese tipo de plataforma de almacenamiento se pueden llevar a cabo
por medio de diferentes paquetes informáticos. Entre otros, QIIME2 (por sus
siglas en inglés: quantitative insights
into microbial ecology version 2), DADA2 (por
sus siglas en inglés: the divisive
amplicon denoising algorithm) u OBITools, que es un conjunto
de programas de Python (lenguaje bioinformático)
desarrollados para simplificar la manipulación de archivos de secuencias (Boyer y col., 2016; Callahan y
col., 2016; Macher y col., 2021).
QIIME2 puede transformar secuencias sin procesar
en diagramas de barras taxonómicas, árboles
filogenéticos, análisis de coordenadas principales y otras
visualizaciones de la diversidad microbiana.
Para DADA2 se incluyen herramientas o procedimientos sobre cómo filtrar tablas
y cómo calcular y visualizar los análisis de ordenamiento y medición de la
diversidad, para lo cual se introdujo un enfoque basado en modelos para
corregir errores del amplicón sin construir OTU. Así, DADA2 es precedido de otro
paquete informático basado en el programa de R, el cual es de código abierto
(https://github.com/benjjneb/dada2) que extiende y mejora el algoritmo de su
predecesor. Mientras que OBITools hace la asignación
taxonómica de secuencias automáticamente contra una base de datos en línea.
Tiene capacidad para filtrar y editar secuencias teniendo en cuenta la
anotación taxonómica y ayuda a establecer líneas
de análisis a medida para una amplia gama de aplicaciones de metabarcoding de DNA, incluidos estudios de
biodiversidad o análisis de dietas.
III. eDNA en agua
III.1. eDNA en acuíferos
El método para obtener eDNA de
acuíferos se efectuó primeramente en ambientes controlados, para observar si
era viable la detección de especies, para posteriormente ser aplicado a
ambientes naturales (Ficetola y col., 2008). El eDNA es, relativamente, una nueva herramienta para el
monitoreo que se utiliza en ambientes acuíferos, ya que las partículas de DNA
se distribuyen homogéneamente en el agua, a comparación del DNA que pudiera
encontrarse en el suelo (Hänfling y col., 2016). La
mayoría de los estudios sobre este tema se enfocan en peces y anfibios, pero
esta herramienta se diversificó tanto que también se usa para identificar mamíferos, reptiles, artrópodos, gasterópodos y bivalvos (Dougherty
y col., 2016).
La técnica del eDNA mostró su utilidad
al ser empleada por primera vez para el monitoreo de peces que habitan la
región de Bacalar (México) y a la vez
detectar especies invasoras que pudieran afectar las playas. Se aplicó
en agua y sedimentos, descubriendo que era más viable en agua, ya que fue capaz
de localizar un mayor número de organismos (Valdez-Moreno y col., 2019). En este estudio se incluyeron 8 sitios de muestreo (1 con 3 localidades), la
mayoría fueron examinadas en más de una ocasión, produciendo un total de 14 eventos de muestreo. Las muestras fueron colocadas en botellas, y tanto éstas
como el equipo de muestreo se manipularon con guantes, para minimizar la
contaminación con DNA humano. En cada sitio se hicieron tres tomas repetidas de
agua y sedimento. Cada muestra se dividió en
dos submuestras de 0.5 L que se filtraron a través de
filtros separados de 0.22 m. Un filtro se almacenó con 1 mL de solución PW1 (solución
encontrada en el estuche comercial DNeasy®
PowerWater® Kit – QIAGEN) con medio de trituración (cuentas que ayudan al molido de la
muestra), mientras que la otra submuestra se colocó
con medio de trituración en un tubo
seco cubierto con papel de aluminio.
III.2. eDNA en ríos
Los ríos son invaluables para los estudios ambientales, ya
que transportan la información genética del
paisaje a lo largo de los tramos que atraviesan, así como los sedimentos,
material orgánico y los nutrientes a su alrededor (Deiner
y col., 2016). El eDNA es empleado para el conteo e
identificación de poblaciones de peces, principalmente, mediante métodos no invasivos,
en contraste con los invasivos usados
anteriormente para esto, donde eran capturados e identificados
morfológicamente, lo que no ayuda mucho a la salud del animal, ya que le provoca estrés y aumenta su depredación (Shaw y
col., 2016).
El eDNA resiste en el agua a
temperatura ambiente y puede cambiar su integridad de unos días a algunas
semanas, de acuerdo a un trabajo de Pont y col. (2018), que reveló patrones cuantitativos de la biodiversidad de peces. También
señaló que en las corrientes de los ríos la concentración de eDNA y su detectabilidad no solo
dependen de las tasas de producción y degradación, sino también de la dilución,
el transporte a través de la red fluvial, la deposición y la resuspensión. Este estudio usó como referencia uno previo de Valentini y col. (2016),
el cual diseñó iniciadores que se probaron exhaustivamente in silico utilizando el programa ecoPCR
(Ficetola y col., 2010) en: (i) una colección de
todas las secuencias de DNA mitocondrial y (ii) en todas las secuencias de DNA disponibles públicamente. El programa ecoPCR es un PCR in silico
que consiste en seleccionar en una base de datos las regiones que coinciden (es
decir, que presentan similitud) con dos iniciadores de PCR. Dichas regiones
deben localizarse en la secuencia seleccionada, de manera que permitan la
amplificación por PCR, lo que fuerza la orientación relativa de las coincidencias
y la distancia entre ellas.
Deiner y
col. (2016), mostraron que el método de eDNA ayudó a
detectar más especies en comparación con un método convencional llamado “kicknet” (que utiliza una red de malla cuadrada con un
mango de poste a cada lado que se emplea para recoger macroinvertebrados
acuáticos en una corriente). Utilizando solo kicknet
se consiguieron de 17 a 24 familias, y combinándolo con eDNA
se obtuvieron de 23 a 40 familias (Deiner y col.,
2016; 2017). Existen otros trabajos reportados en Indiana, EE. UU., donde el
método combinado ayudó a identificar especies que otros métodos pasaron por
alto, en comparación con el método convencional (kicknet).
Deiner y col. (2016; 2017) identificaron un promedio
de 12 especies, entre las que se encontraron Semotilus atromaculatus, Lepomis cyanellus, Etheostoma nigrum, Cottus bairdii, Rhinichthys obtusus, Catostomus
commersonii, Oncorhynchus
mykiss, Salmo trutta,
Amblopites rupestris,
Micropterus dolomieu,
Lepomis macrochinus,
y Etheostoma caeruleum;
y con kicknet y eDNA se obtuvieron 4 especies más; Ameiurus natalis, Cyprinus carpio, Umbra pygmaea
y Micropterus salmoides,
dando un promedio de 16 especies en 2013 (Olds
y col., 2016; Valdez-Moreno y col., 2019).
Finalmente, otro estudio en Japón usando eDNA,
descubrió especies acuáticas raras, en peligro de extinción, y especies
invasoras (Yamamoto y col., 2017).
IV. eDNA
en ambientes terrestres
Los sedimentos son una fuente muy valiosa para el estudio de eDNA,
el DNA puede permanecer desde periodos cortos hasta miles de años,
dependiendo de las condiciones del ambiente
donde se encuentre la muestra (Buxton y col., 2018). Algunos estudios demuestran que el
eDNA se mantiene más tiempo en el sedimento que en la
superficie del agua. Turner y col. (2015) demostraron que el DNA de la carpa
asiática de cabeza grande se conserva por 4 meses en el sedimentos y está más concentrado
que en las columnas de agua (Turner y col., 2015).
Así como en el agua, los sedimentos
terrestres son muy importantes, ya que ayudan a investigar y reconstruir
evidencias de animales o comunidades de plantas que habitaron la tierra hace miles de años (Deiner
y col., 2017). Es decir, la fauna y la flora ancestrales dejaron ahí
rastros de DNA extracelular. En 2013, otro trabajo estudió sedimentos en
Siberia y en Nueva Zelanda, descubriendo que
contenían DNA de animales extintos, como el mamut lanudo, en el primer
sitio, y las aves de Moa en el segundo (Pedersen y col., 2015). Para lograr el metabarcoding se utilizaron iniciadores genéricos (o
universales), que están diseñados para apuntar a varios taxones simultáneamente,
entre ellos, plantas, animales o muestras fecales, en contraste con los
iniciadores específicos, diseñados para amplificar sólo unas pocas especies seleccionadas. Por esta razón y derivado, que con la tecnología molecular podía detectarse DNA, los investigadores le llamaron
DNA ancestral o “antiguo” (aDNA, por sus siglas en
inglés: ancient DNA).
Aunque parece “fácil” la detección del aDNA,
se debe considerar que su campo está plagado de obstáculos metodológicos
significativos que incluyen, entre otros, daño post mortem a biomoléculas conservadas, contaminación de
muestras y reactivos por DNA moderno y la presencia de inhibidores de
reacciones enzimáticas (McHugo y col., 2019). En
general, diversos factores que pueden comprometer irrevocablemente la autenticidad
y reproducibilidad de un DNA amplificado a
partir de muestras arqueológicas o fortuitas encontradas en zonas donde
posiblemente habitaron animales o plantas prehistóricas. Sin embargo, con los
cuidados de muestreo, transportación, enriquecimiento
del DNA en el momento de su extracción
y, claro, la tecnología de la amplificación, se tiene el potencial para
estudios de detección de flora y fauna ancestral.
V. eDNA en el aire
El agua y el aire son capaces de desplazar el DNA ambiental
más rápido y a mayores distancias (Barnes y Turner, 2016). Específicamente, el
aire es un medio importante de transporte de microorganismos ya que supera barreras geográficas (Yooseph
y col., 2013) y traslada diferentes
partículas de material diverso, polvo, microorganismos, esporas, polen,
entre otros.
Por ello, el eDNA puede tener
aplicaciones en la salud humana utilizando
como muestra el aire ambiental de
alguna zona o ecosistema dado. Determinar el microbioma
en el aire puede influir en las decisiones de salud con respecto a los alérgenos (partícula o microorganismos que genera una reacción inmunológica de
hipersensibilidad), los microorganismos patógenos
y la contaminación del aire. Así se debe considerar, por ejemplo, la
actual pandemia del síndrome respiratorio agudo grave (SARS, por sus siglas en
inglés: severe acute respiratory syndrome), la cual es
una enfermedad respiratoria viral causada por un coronavirus del tipo 2, llamado
coronavirus asociado al SARS (SARS-CoV-2) que provoca la enfermedad conocida
como COVID-19 (Wang y col., 2020). Para el caso de los microorganismos en aire,
la identificación actualmente se realiza mediante la observación microscópica,
la cual se torna laboriosa y en muchos casos no se logran reconocer, a nivel de
género, todas las especies relevantes (Kraaijeveld y
col., 2015) o cultivables, para su posterior identificación. Mediante la recolección de muestras del aire, aplicación del metabarcoding y la tecnología NGS es
posible determinar la composición del microbioma en el
aire y sus posibles implicaciones para la salud humana y el ecosistema.
Si bien, el uso más frecuente de metabarcoding
ambiental es en entornos naturales, también
se puede aplicar en interiores. En particular, los microorganismos
transportados por el aire son una preocupación crítica en los espacios de atención
a la salud, ya que algunos pueden causar infecciones asociadas a los hospitales
y, en general, a lugares donde se tienen en uso equipos de aire acondicionado que pueden contener bacterias, virus u hongos (Cao y col., 2014;
Banchi y col.,
2018).
Un ejemplo de la aplicación de esta metodología fue en 2015,
cuando se realizó un estudio en aguas marinas para la identificación de orcas (Orcinus orca); para ello se tomaron muestras de agua y aire/superficie de las Islas de
San Juan. Del aire/superficie se obtuvo la
muestra de DNA por medio de filtros. El análisis de las muestras de las
superficie encontró excelente concentración de DNA y se logró secuenciar a los microorganismos (Baker y col., 2018).
Para este estudio, además del eDNA, se utilizó una variante del PCR llamado PCR por gotitas digitales
(ddPCR, por sus siglas en inglés: droplet-digital
PCR), la cual es una nueva y poderosa tecnología para cuantificar niveles bajos
de DNA mediante el fraccionamiento de una reacción de PCR en más de 20 000
gotas utilizando una emulsión de aceite (Doi y col., 2015).
En otro caso, se comprobó que se puede
identificar el polen de las plantas que potencialmente es alérgeno. Lo que se
hizo fue filtrar las muestras de aire (Kraaijeveld y
col., 2015) para localizar el tipo de polen.
En realidad, la utilidad del metabarcoding de eDNA se
vuelve infinita, en virtud de que es posible hacer un procedimiento de análisis relativamente
sencillo, aislando un fluido (aire o agua)
de uno o de diferentes entornos, y después
determinando uno o varios organismos, con el examen del microbioma de un
ambiente específico; en muchos casos, sin necesidad de crecimiento o siembra de tales microorganismos.
CONCLUSIONES
El avance de la ciencia conlleva a nuevos descubrimientos, técnicas
y metodologías producto del trabajo de investigación, dedicación y tiempo. En
biología molecular abarca nuevas maneras de analizar lo que antes era casi imposible,
debido a que no se podían identificar simultáneamente a todos o una parte de
los organismos de un ambiente dado. La reciente introducción de la técnica metabarcoding
de ácido desoxirribonucleico (DNA) y en particular del estudio de DNA ambiental (eDNA)
ha permitido alcanzar ese objetivo. Actualmente existe información
disponible sobre el metabarcoding y eDNA, en particular, en agua, lo cual es muy relevante para
la detección de especies nuevas, previamente descritas o especies invasoras
para el ecosistema, los humanos, los animales
o las plantas. Sin embargo, aún faltan más estudios en otros tipos de muestras,
como suelos y aire. Se espera que en un futuro esta sea la manera para la
caracterización y clasificación de especies y que su uso se generalice para el
monitoreo de microorganismos, especies terrestres o acuáticas que se encuentran
en números muy bajos, se cree que ya están
extintas o son especies nuevas. Es importante destacar que se requiere
de técnicas especiales, como la reacción en cadena de la polimerasa (PCR) y
secuenciación de terminación de la cadena de Sanger o
de siguiente generación. En esta primera etapa es necesario generar las bases
de datos de todo tipo de especímenes, por ejemplo, de cuerpos de fluidos, para que existan referencias con qué compararse
o en su defecto, si ya existen, comparar las secuencias obtenidas con esas
bases de datos previamente creadas. Finalmente, con el metabarcoding
se generará un código de barras para algún
tipo de muestra que se desee estudiar y con esto lograr el inventario de
la biodiversidad de un área en particular y por qué no, el inventario total de la Tierra, entre otras aplicaciones mencionadas en este trabajo.
Referencias
Adams, C. I., Knapp, M., Gemmell, N.
J., Jeunen, G. J., Bunce, M., Lamare, M. D., and
Taylor, H. R. (2019). Beyond Biodiversity: Can Environmental DNA (eDNA) Cut It as a Population Genetics Tool? Genes.
10(3): 192.
Andruszkiewicz, E. A., Sassoubre, L. M.,
and Boehm, A. B. (2017a). Persistence of marine fish environmental DNA and the
influence of sunlight. PLoS One. 12(9).
Andruszkiewicz, E. A., Starks, H. A., Chavez, F. P., Sassoubre, L. M., Block, B. A., and Boehm, A. B. (2017b).
Biomonitoring of marine vertebrates in Monterey Bay using eDNA metabarcoding. PLoS One. 12(4): e0176343.
Austin, B. (2017). The value of
cultures to modern microbiology. Antonie
Van Leeuwenhoek. 110(10): 1247-1256. Banchi, E., Ametrano, C. G., Stanković, D., Verardo, P., Moretti, O., Gabrielli,
F., …, and Muggia, L.
(2018). DNA metabarcoding uncovers fungal diversity
of mixed airborne samples in Italy. PLoS
One. 13: e0194489.
Baker, C. S., Steel, D., Nieukirk, S., and Klinck, H.
(2018). Environmental DNA (eDNA)
from the wake of the whales: Droplet digital PCR for detection and
species identification. Frontiers in Marine Science. 5: 1-11.
Barnes, M. A. and Turner, C. R.
(2016). The ecology of environmental DNA and implications for conservation genetics.
Conservation Genetics. 17(1): 1-17.
Benson, D. A., Cavanaugh, M., Clark,
K., Karsch-Mizrachi, I., Lipman, D. J., Ostell, J., and Sayers, E. W. (2013). GenBank. Nucleic
Acids Research. 41(D1): D36-D42.
Bortolus, A. (2008).
Error cascades in the biological sciences: The unwanted consequences of using bad taxonomy in ecology. AMBIO:
A Journal of the Human Environment. 37(2):
114-118.
Boyer, F., Mercier, C., Bonin, A.,
Le-Bras, Y., Taberlet, P., and Coissac,
E. (2016). Obitools: a unix-inspired
software package for DNA metabarcoding. Molecular Ecology Resources.
16(1): 176-182.
Buxton, A. S., Groombridge,
J. J., and Griffiths, R. A. (2018). Seasonal variation in environmental DNA
detection in sediment and water samples. PLoS
One. 13(1): 1-14.
Bylemans, J., Gleeson, D. M., Hardy, C. M., and Furlan,
E. (2018). Toward an ecoregion scale evaluation of eDNA
metabarcoding primers: A case study for the
freshwater fish biodiversity of the Murray-Darling Basin (Australia). Ecology
and Evolution. 8(17): 8697-8712.
Callahan, B. J., McMurdie,
P. J., Rosen, M. J., Han, A. W., Johnson, A. J., and Holmes, S. P. (2016).
DADA2: High-resolution sample inference from Illumina amplicon data. Nature
Methods. 13(7): 581-583.
Cao, C., Jiang, W., Wang, B., Fang,
J., Lang, J., Tian, G., …, and Zhu, T. F. (2014). Inhalable microorganisms in Beijing’s
PM2.5 and PM10 pollutants during a severe smog event. Environmental Science
& Technology. 48(3): 1499-1507.
Cowart, D. A., Murphy, K. R., and
Cheng, C. H. C. (2018). Metagenomic sequencing of environmental DNA reveals marine
faunal assemblages from the West Antarctic Peninsula. Marine Genomics.
37: 148-160.
Creer, S., Deiner, K., Frey, S., Porazinska, D., Taberlet, P.,
Thomas, W., …, and Bik, H. (2016). The ecologist’s
field guide to sequence-based identification of biodiversity. Methods in Ecology and Evolution. 7(9): 1008-1018.
Deiner, K., Bik, H. M., Mächler,
E., Seymour, M., Lacoursière-Roussel, A., Altermatt, F., …, and Bernatchez, L. (2017). Environmental DNA metabarcoding:
Transforming how we survey animal and plant communities. Molecular Ecology.
26(21): 5872-5895.
Deiner, K., Fronhofer, E. A., Mächler, E., Walser, J. C., and Altermatt,
F. (2016). Environmental DNA reveals that rivers are conveyer belts of
biodiversity information. Nature Communications. 7(1): 1-9.
Díaz-Ferguson, E.
E. and Moyer, G. R. (2014). History, applications,
methodological issues and perspectives for the use environmental DNA (eDNA) in marine and freshwater
environments. Revista de Biología Tropical. 62(4): 1273-1284.
DOF, Diario Oficial de la Federación (2020). Ley de Aguas Nacionales.
[En línea]. Disponible en: http://www.diputados.gob.mx/LeyesBiblio/pdf/16_060120.pdf.
Fecha de consulta: 10 de junio de 2021.
Doi, H., Uchii,
K., Takahara, T., Matsuhashi,
S., Yamanaka, H., and Minamoto, T. (2015). Use of droplet digital PCR for
estimation of fish abundance and biomass in environmental DNA surveys. PLoS One. 10(3):
e0122763.
Dougherty, M. M., Larson, E. R.,
Renshaw, M. A., Gantz, C. A., Egan, S. P., Erickson,
D. M., and Lodge, D. M. (2016). Environmental
DNA (eDNA) detects the invasive rusty crayfish Orconectes rusticus at low
abundances. Journal of Applied Ecology. 53(3): 722-732.
Epp, L. S., Boessenkool,
S., Bellemain, E. P., Haile, J., Esposito, A., Riaz,
T., …, and Brochmann, C.
(2012). New environmental metabarcodes
for analysing soil DNA: potential for studying
past and present ecosystems. Molecular Ecology. 21: 1821-1833.
Evans, N. T., Olds, B. P., Renshaw,
M. A., Turner, C. R., Li, Y., Jerde, C. L., ..., and
Lodge, D. M. (2016). Quantification of mesocosm fish and amphibian species
diversity via environmental DNA metabarcoding. Molecular Ecology Resources.
16(1): 29-41.
Ficetola, G. F., Coissac, E., Zundel,
S., Riaz, T., Shehzad, W., Bessière, J., ..., and Pompanon, F. (2010). An
in silico approach for the evaluation of DNA
barcodes. BMC Genomics: 11(1): 434.
Ficetola, G. F., Miaud, C., Pompanon, F., and Taberlet, P.
(2008). Species detection using environmental DNA from water samples. Biology
Letters. 4(4): 423-425.
Gibson, J. F., Shokralla,
S, Curry, C., Baird, D. J., Monk, W. A., King, I., and Hajibabaei,
M. (2015). Large-Scale Biomonitoring of Remote and Threatened Ecosystems via
High-Throughput Sequencing. PLoS One.
10(10): e0138432.
Green, M. R. and Sambrook, J. (2017).
Isolation of High-Molecular-Weight DNA Using Organic Solvents. Cold Spring Harb Protoc. (4):
pdb.prot093450.
Hänfling, B., Handley, L. L., Read, D. S., Hahn, C., Li, J.,
Nichols, P., …, and Winfield, I. J. (2016).
Environmental DNA metabarcoding of lake fish communities reflects long-term
data from established survey methods. Molecular Ecology. 25(13):
3101-3119.
Hering, D., Borja, A., Jones, J. I., Pont, D., Boets, P., Bouchez, A., …, and Kelly, M. (2018). Implementation options for DNA-based identification into ecological
status assessment under the European Water Framework Directive. Water
Research. 138: 192-205.
Hernández-González, J., Inza, I., and Lozano, J. A. (2016). Weak supervision and other non-standard classification problems: A taxonomy. Pattern
Recognition Letters. 69: 49-55.
Ivanova, N.,
Valdez-Moreno, M., and Elias-Gutierrez, M. (2019). Resident or Invasive Species? Environmental DNA Can Provide
Reliable Answers. IBOL Barcode Bulletin. 9(1): 9-11.
Kraaijeveld, K., de-Weger, L. A., Ventayol-García, M., Buermans, H., Frank, J., Hiemstra, P. S., and den-Dunnen, J. T. (2015). Efficient and sensitive
identification and quantification of airborne pollen using
next-generation DNA sequencing. Molecular Ecology Resources. 15(1):
8-16.
Koch, J., Gantenbein,
S., Masania, K. Stark, W. J., Erlich,
Y., and Grass, R. N. (2020). A DNA-of-things storage architecture to create materials with embedded memory. Nature
Biotechnology. 38(1): 39-43.
Koslowski, M., Moran, D.,
Tisserant, A., Verones, F.,
and Wood, R. (2020). Quantifying Europe’s
biodiversity foot-prints and the role of urbanization and income. Global
Sustainability. 3: E1.
Lacoursière-Roussel, A., Dubois, Y., Normandeau,
E., and Bernatchez, L. (2016). Improving
herpetological surveys in eastern North
America using the environmental DNA method1. Genome. 59(11): 991-1007.
Liu, Q., Zhang, Y., Wu, H., Liu, F.,
Peng, W., Zhang, X., ..., and Zhang, H. (2020). A
Review and Perspective of eDNA Application to Eutrophication and HAB Control in
Fresh-water and Marine Ecosystems. Microorganisms. 8(3): 417.
Macher, T. H., Beermann,
A. J., and Leese, F. (2021). Taxon TableTools: A
comprehensive, platform-independent graphical user
interface software to explore and visualise DNA metabarcoding data. Molecular Ecology Resources. 21(5):1705-1714.
Mani, I. (2020). Current Status and
Challenges of DNA Sequencing. In V. Singh (Ed.), Advances in Synthetic
Biology (pp. 71-80). Alemania: Springer,
Singapore.
McHugo, G. P., Dover, M. J., and MacHugh,
D. E. (2019). Un locking the origins and biology of
domestic animals using ancient DNA and paleogenomics.
BMC Biology. 17(1): 1-20.
Muha,
T. P., Robinson, C. V., Garcia-de-Leaniz,
C., and Consuegra, S. (2019). An optimised eDNA protocol for detecting fish in lentic and lotic freshwaters
using a small water volume. PLoS One.
14(7): e0219218.
Olds, B. P., Jerde,
C. L., Renshaw, M. A., Li, Y., Evans, N. T., Turner, C. R., …, and Lamberti, G. A. (2016). Estimating species
richness using environmental DNA. Ecology and Evolution. 6(12):
4214-4226.
Pedersen, M. W., Overballe-Petersen, S., Ermini,
L., DerSarkissian, C., Haile, J., Hellstrom,
M., ..., and Willerslev, E. (2015). Ancient and modern environmental DNA. Philosophical Transactions of the Royal Society B: Biological
Sciences. 370(1660):
20130383.
Pizarro, O. R. (2003). Proyecto de ley para un registro de ADN de utilización Criminalística. Revista Conamed. 8(2): 21-34.
Pont, D., Rocle,
M., Valentini, A., Civade,
R., Jean, P., Maire, A., …, and Dejean,
T. (2018). Environmental DNA reveals quantitative patterns of fish biodiversity
in large rivers despite its downstream transportation. Scientific Reports.
8(1): 1-13.
Ratnasingham, S. (2019). mBRAVE: The Multiplex Barcode Research And Visualization Environment. Biodiversity Information
Science and Standards. 3.
Raven, P. H. (2020) Biodiversity: A
Global Perspective. In G.Dar and A. Khuroo (Eds.), Biodiversity of the Himalaya: Jammu and
Kashmir State. Topics in Biodiversity and Conservation (pp. 156-180). Alemania: Springer, Singapore.
Rimieri, P. (2017). Genetic Diversity and Genetic Variability: Two Different Concepts Associated To
Plant Germplasm and Breeding. Journal of Basic and Applied Genetics.
28(2): 7-13.
Ruppert, K. M.,
Kline, R. J., and Rahman, M. S. (2019). Past, present, and future perspectives of environmental DNA (eDNA) metabarcoding: A systematic review
in methods, monitoring, and
applications of global eDNA. Global Ecology and Conservation. 17:
e00547.
Salipante, S. J. and Jerome, K. R. (2020). Digital PCR—An Emerging Technology with Broad Applications in
Microbiology. Clinical Chemistry. 66(1): 117-123.
Sanger, F.,
Nicklen, S., and Coulson, A. R. (1977). DNA sequencing with chain-terminating inhibitors. Proceedings of the National Academy of Sciences of the United
States of America. 74(12):
5463-7.
Scheffers, B., Joppa, L.,
Pimm, S., and Laurance, W. (2012). What we know and don’t know about Earth’s missing
biodiversity. Trends in Ecology &
Evolution. 27(9): 501-10.
Shaw, J. L. A., Clarke, L. J., Wedderburn,
S. D., Barnes, T. C., Weyrich, L. S., and Cooper, A.
(2016). Comparison of environmental DNA
metabarcoding and conventional fish survey methods in a river system. Biological
Conservation. 197: 131-138.
Soliman, T., Yang, S. Y., Yamazaki,
T., and Jenke-Kodama, H. (2017). Profiling soil
microbial communities with next-generation
sequencing: the influence of DNA kit selection and technician technical expertise.
PeerJ. 5: e4178.
Taberlet, P., Coissac, E., Hajibabaei, M., and Rieseberg, L.
H. (2012). Environmental DNA. Molecular Ecology. 21(8): 1789-1793.
Thomsen, P. F., Møller,
P. R., Sigsgaard, E. E., Knudsen, S. W., Jørgensen, O. A., and Willerslev,
E. (2016). Environmental DNA from Seawater Samples Correlate with Trawl Catches
of Subarctic, Deepwater Fishes. PLoS One.
11(11): e0165252.
Thomsen, P. F. and Willerslev, E. (2015). Environmental DNA - An emerging tool
in conservation for monitoring past and present biodiversity. Biological Conservation.
183: 4-18.
Turner, C. R., Uy,
K. L., and Everhart, R. C. (2015). Fish environmental DNA is more concentrated
in aquatic sediments than surface water. Biological Conservation. 183:
93-102.
UNEP, United
Nations Environment Programme (2011). Convention of
Biological Diversity: Strategic Plan for Biodiveristy 2011-2020. [En línea]. Disponible en: https://www.cbd.int/doc/strategic-plan/2011-2020/Aichi-Targets-EN.pdf. Fecha
de consulta: 21 de enero de 2021.
Ushio,
M., Fukuda, H., Inoue, T., Makoto, K., Kishida, O., Sato, K., ..., and Miya, M. (2017). Environmental DNA enables detection of terrestrial
mammals from forest pond water. Molecular Ecology Resources.
17(6): e63-e75.
Valdez-Moreno, M., Ivanova, N. V.,
Elías-Gutiérrez, M., Pedersen, S. L., Bessonov, K., and
Hebert, P. D. (2019). Using Edna to biomonitor the fish community in a tropical oligotrophic
lake. PLoS
One. 14(4): 1-22.
Valentini, A., Taberlet, P., Miaud, C., Civade, R., Herder,
J., Thomsen, P. F., …, and Dejean,
T. (2016). Next-generation monitoring of
aquatic biodiversity using environmental DNA metabarcoding. Molecular
Ecology. 25(4): 929-942.
van-Dijk, E. L, Jaszczyszyn, Y.,
Naquin, D., and Thermes, C. (2018). The Third
Revolution in Sequencing Technology. Trends in
Genetics. 34(9): 666-681.
Vázquez-Ramos, J. (2016). Reparación del ADN: un asunto de
vida... y de Premios Nobel. Educación Química. 27(2): 93-96.
Wang, M. Y., Zhao, R., Gao, L. J., Gao, X. F., Wang, D. P., and Cao, J. M. (2020).
SARS-CoV-2: Structure, Biology, and Structure-Based
Therapeutics Development. Frontiers in Cellular and Infection
Microbiology. 10: 587269.
Wang, S., Yan, Z., Hänfling, B., Zheng, X., Wang, P., Fan, J., and Li, J. (2021). Methodology of fish eDNA and its applications in ecology and environment. Science of the Total Environment.
755: 142622.
Yamamoto, S.,
Masuda, R., Sato, Y., Sado, T., Araki, H., Kondoh, M., ..., and
Miya, M. (2017). Environmental DNA metabarcoding reveals local fish communities
in a species-rich coastal sea. Scientific Reports. 7(1): 1-12.
Yildiz, I., Açıkkalp, E., Caliskan, H., and Mori, K. (2019). Environmental pollution cost analyses of biodiesel and diesel fuels for a diesel engine. Journal of
Environmental Management. 243: 218-226.
Yooseph, S., Andrews-Pfannkoch, C.,
Tenney, A., McQuaid, J., Williamson, S., Thiagarajan,
M., …, and Venter, J. C. (2013). A metagenomic
framework for the study of airborne microbial communities. PLoS
One. 8(12): e81862.
Zhu, H., Zhang, H., Xu, Y., Laššáková, S., Korabečná, M., and
Neužil, P. (2020). PCR past, present and
future. Biotechniques.
69(4): 317-325.