https://doi.org/10.29059/cienciauat.v17i2.1728
Calidad
microbiológica: detección de Aeromonas sp y Pseudomonas sp en garrafones provenientes de
pequeñas plantas purificadoras de agua
Microbiological quality:
detection of Aeromonas sp and Pseudomonas sp
in jugs from small water purification establishments
Aeromonas y Pseudomonas en
agua purificada
Berenice Venegas, Marco
Antonio Tello-Hernández, Verónica Cepeda-Cornejo, Dalia Molina-Romero*
*Correspondencia: dalia.molina@correo.buap.mx/Fecha
de recepción: 9 de agosto de 2022/Fecha de aceptación: 9 de noviembre de 2022/Fecha
de publicación: 31 de enero de 2023.
Benemérita Universidad
Autónoma de Puebla, Facultad de Ciencias Biológicas, Ciudad Universitaria,
boulevard Valsequillo y Av. San Claudio, Ed. BIO1, colonia Jardines de San
Manuel, Puebla, Puebla, México, C. P. 72592.
Resumen
Las plantas purificadoras de agua que carecen de un
adecuado sistema de control de calidad pueden generar problemas de salud
pública. El objetivo de este estudio fue examinar la calidad microbiológica del
agua proveniente de pequeñas plantas purificadoras de la ciudad de Puebla, así
como, determinar la existencia de bacterias Aeromonas sp y Pseudomonas sp,
y caracterizar si presentan un fenotipo patógeno oportunista. Se recolectaron
70 muestras de garrafones de agua de 25 establecimientos. La cuantificación
bacteriana se realizó mediante el método de goteo en placa. Se comprobaron los
géneros microbianos mediante análisis bioquímico. En las cepas que mostraron
discrepancia se utilizó la identificación molecular con base a secuencias
parciales del gen 16S rRNA para confirmar su especie
y se les evaluaron sus características de patogenicidad: multirresistencia
a antibióticos, producción de biopelícula y actividad hemolítica. El 40 % de
las plantas purificadoras no cumplieron con la calidad microbiológica del agua
para consumo humano. El 41.4 % de los garrafones de agua muestreados incumplió
la normativa, presentando coliformes totales 35.7 %, Pseudomonas 30
%, Enterococcus faecalis 8.6
% y bacterias coliformes fecales el 5.7 %. Se obtuvieron 56 aislados,
provenientes de los 29 garrafones contaminados; 10 de ellos se caracterizaron
molecularmente, resultando 7 aislados relacionados con especies diferentes de
P. aeruginosa y 3 con especies de Aeromonas.
De los aislados de Pseudomonas, 5 presentaron resistencia a 2
familias de antibióticos y 2 mostraron multirresistencia.
El 36 % de los 10 aislados produjeron hemólisis y biopelícula. Dos cepas
de Aeromonas mostraron resistencia a
Cefalosporina 3ª generación pero no produjeron
hemólisis. Los 10 aislados analizados fueron clasificados como no patógenos. Es
necesario un seguimiento sanitario más estricto para lograr el cumplimiento de
las normas nacionales e internacionales relacionadas con el consumo de agua
purificada, para evitar dañar la salud de los consumidores.
Palabras clave: Coliformes, Aeromonas, Pseudomonas, biopelícula, multirresistencia.
Abstract
Water
purification establishments that lack an adequate quality control system can
cause public health problems. The objective of this study was to examine the
microbiological quality of water from small purification establishments in the
city of Puebla, as well as to determine the existence of Aeromonas sp
and Pseudomonas sp bacteria, and to characterize whether they present an
opportunistic pathogenic phenotype. 70 water jug samples were collected from 25
establishments. Bacterial quantification was performed using the drop plate
method. Microbial genera were determined by biochemical analysis using the
standard methodology. In the strains that showed discrepancy, molecular
identification based on partial sequences of the 16S rRNA gene was used to
confirm their species, and their pathogenic characteristics were evaluated: multiresistance to antibiotics, biofilm production, and
hemolytic activity. The results showed that 40 % of the purification
establishments did not comply with the microbiological quality of water for
human consumption. Similarly, 41.4 % of the jugs of water sampled failed to
comply with the regulations, presenting total coliforms 35.7 %, Pseudomonas
30 %, Enterococcus faecalis 8.6 % and fecal coliform bacteria 5.7 %.
Likewise, 56 isolates were obtained from the 29 contaminated jugs, of which 10
were molecularly characterized, resulting in 4 different species for P.
aeruginosa and 3 for Aeromonas. Of the 7 Pseudomonas
isolates, 5 presented resistance to 2 families of antibiotics and 2 showed multiresistance. In total, 36 % of the 10 isolates produced
hemolysis and biofilm. Two Aeromonas strains showed resistance to 3rd
generation Cephalosporin but did not produce hemolysis. The 10 isolates
analyzed were classified as non-pathogenic. A stricter sanitary monitoring is
necessary to achieve compliance with national and international standards
related to the consumption of purified water, to avoid harming the health of
consumers.
Keywords: Coliforms, Aeromonas,
Pseudomonas, biofilm, multiresistance.
Introducción
El agua es esencial para los procesos biológicos y el
mantenimiento de los ecosistemas, por lo que es importante para los organismos
vivos (Pichel y col., 2019). Son varios los factores que afectan la calidad del
agua, siendo los más notables las actividades antropogénicas en los asentamientos
urbanos, el aumento de la industrialización y la generación de desechos (Ji y col.,
2021; OMS, 2022), que contribuyen a la presencia de agentes infecciosos,
químicos tóxicos y radiaciones en el agua de acuerdo al
Diatrio Oficial de la Federación (DOF, 2015b). La contaminación
del agua es un problema de salud púbica y su consumo puede causar enfermedades gastrointestinales
que pueden llevar a la muerte, como gastroenteritis hemorrágica, cólera y
diarrea aguda (Gutiérrez-Del-Río y col., 2018). Las bacterias coliformes causan
estas enfermedades (Loyola y col., 2020).
Las
infecciones diarreicas provocan alta morbilidad
tanto en países desarrollados como en vías de desarrollo, debido a su alta
incidencia en la población de niños menores de cinco años y adultos mayores (Saxena y col., 2015; OMS, 2017). Este tipo de
enfermedades representa, para ambos grupos de naciones, un alto costo económico
a nivel de salud e impacto social (Kamal y Abdel-Latef, 2015; Sacchetti y col., 2015).
Las
bacterias comúnmente consideradas agentes etiológicos de la diarrea aguda son
los diferentes patotipos de Escherichia
coli, Salmonella sp,
Campylobacter sp, Yersinia
sp, Vibrio cholerae y Enterococcus faecalis
(Wu y col., 2011; DOF, 2015b; DOF,
2021).
También se han identificado otros géneros como
patógenos oportunistas que causan diarrea en personas inmunocomprometidas, como
Pseudomonas sp, Klebsiella
sp y Aeromonas sp.
Estas bacterias han sido aisladas del agua potable, de acuerdo con el Diario
Oficial de la Federación (DOF, 2015b). Así
mismo, Aeromonas es capaz de sobrevivir
en agua que ha sido clorada, y su presencia se ha asociado con enfermedades gastrointestinales en humanos y animales (Skwor y col., 2014; Miyagi y col.,
2017).
La
patogenicidad de un microorganismo es la habilidad de causar daño a un
hospedero (Madigan y col., 2015), una bacteria
patógena emplea diferentes factores de virulencia para dañar y adaptarse contra
los diferentes mecanismos de defensa del hospedero (Jurado-Martín y col., 2021). La formación de biopelícula confiere virulencia
en bacterias patógenas oportunistas, ya que ayuda a la adhesión y a la colonización bacteriana, reduce la sensibilidad
a los antibióticos y evita que el sistema inmunológico reconozca a la bacteria
(Chenia y Duma,
2017). La multirresistencia es definida como la resistencia al menos a un antibiótico de tres o
más familias diferentes, esto favorece a las bacterias patógenas para
continuar con la infección en su hospedero (Madigan y
col., 2015), sin ser eliminado aún con el suministro
de antibióticos. Estas bacterias aumentan la morbilidad y la mortalidad en la población (Nath y col.,
2020). La hemólisis es considerada un factor de virulencia, debido a que la bacteria produce y transporta
proteínas y toxinas que destruyen al
eritrocito, la hemoglobina y otras células (Madigan
y col., 2015).
Aeromonas y Pseudomonas
han sido descritas como bacterias multirresistentes a antibióticos y formadoras
de biopelícula (Govender y col., 2021); algunas
especies de estos géneros bacterianos se han reportado con capacidad hemolítica
(Nowrotek y col., 2021). La biopelícula es una estructura
constituida por exopolisacáridos y bacterias, que le
otorga a la comunidad bacteriana protección ante el estrés ambiental (Sala-Comorera
y col., 2016). En el caso de las bacterias patógenas, la biopelícula le
confiere atributos para el establecimiento de la infección al hospedero,
además, que es un ambiente idóneo que favorece la transferencia horizontal de genes
que codifican la resistencia a antibióticos. La presencia de estas bacterias en
el agua puede ser una fuente de diseminación de dichos genes de multirresistencia entre los miembros de la comunidad bacteriana
(Mulamattathil y col., 2014).
Las Naciones Unidas se han encargado de generar políticas
de prevención para reducir las enfermedades
gastrointestinales provocadas por la
contaminación del agua (Cerna-Cortes y col., 2019). La Organización
Mundial de la Salud (OMS, 2022) es
responsable de desarrollar estándares
internacionales para cumplir con los requisitos que garantizan la
calidad del agua y promover la salud de las poblaciones (DOF, 2015c; Pichel y col., 2019). En México, las Normas
Oficiales Mexicanas establecidas por el gobierno federal, regulan el
saneamiento del agua para consumo
personal y doméstico. Estos lineamientos establecen y describen los límites
permisibles de los indicadores
bacteriológicos, con el fin de
asegurar y preservar la calidad del agua
que se entrega al consumidor (DOF, 2021): 1) coliformes fecales y totales (DOF, 2015c), 2) Pseudomonas aeruginosa (DOF, 2015b), 3) Enterococcus
faecalis (DOF, 2015b), 4) Vibrio cholerae (Loyola y col., 2020), y 5) esporas de Clostridium reductoras de sulfito (Miyagi y col.,
2017); asimismo, determinan los análisis a los que debe someterse el agua para
evaluar el control sanitario del agua (DOF, 2015a).
En México, existen procesos de potabilización de agua en la red municipal de agua que cumplen con los estándares establecidos; sin embargo, los
consumidores han optado por obtener agua de establecimientos dedicados a la
purificación y embotellamiento debido a su desconfianza hacia los sistemas
de abastecimiento, sus métodos y
características organolépticas del agua local (DOF, 2015b; DOF, 2021). Aunque,
se tiene evidencia que cuestiona el cumplimiento de las normas sanitarias por
parte de establecimientos dedicados a la purificación y venta de agua para consumo humano, particularmente de las plantas más pequeñas. La mala calidad de
agua que comercializan estas empresas (Aziz y col.,
2017; Mohamed y col., 2020), es consecuencia del incumplimiento de las normas
sanitarias (DOF, 2015b), o el mal uso de etiquetas y sellos de garantía de
empresas que si cumplen con los estándares solicitados (Pichel y col.,
2019).
Los
estudios de la calidad del agua realizados a pequeñas plantas potabilizadoras,
se han enfocado en grandes ciudades,
como la Ciudad de México (Cerna-Cortes y col., 2019); sin considerar la
producción de biopelícula y la multirresistencia a
antibióticos; además de la identificación del género Aeromonas
y especies del género Pseudomonas
distintas a P. aeruginosa.
El objetivo de este estudio fue analizar la calidad del
agua proveniente de pequeñas plantas purificadoras de la ciudad de Puebla, en
función de los indicadores bacteriológicos (coliformes fecales y totales, P.
aeruginosa y E. faecalis)
establecidos en las normas nacionales e
internacionales; así mismo, identificar a las bacterias que presenten un
fenotipo patógeno oportunista, mediante la
determinación de multirresistencia a
antibióticos, la capacidad de hemólisis y la
formación de biopelícula.
MATERIALES Y MÉTODOS
Colecta de la muestra
La ciudad de Puebla se encuentra en la parte centro
occidental del estado de Puebla, México (Longitud 98°17’38.76” W 98°01’12.72”
W, Latitud 18°50’12.48” N 19°13’51.24” N). Presenta un clima predominante
subhúmedo y templado, con lluvias de verano de acuerdo a
información del Instituto Nacional de Estadística y
Geografía (INEGI, 2010).
Las muestras de agua (n = 70) fueron recolectadas a
partir de 25 plantas purificadoras de pequeña escala, localizadas en la zona
norponiente (NP) y suroriente (SO) en la ciudad de Puebla. Durante el periodo
de marzo a noviembre de 2019, se realizó la compra de al menos 1 garrafón de agua de 20 L por semana, proveniente
de 1 de las 25 purificadoras pequeñas, hasta completar dos muestras en cada
establecimiento (n = 50). Únicamente en 10 establecimientos se compraron dos
muestras adicionales (n = 20), debido a la detección de contaminación microbiana.
Se verificó que los garrafones estuvieran bien cerrados y con el sello de garantía
del proveedor. El día de la compra, a cada garrafón se le quitó el empaque y se
tomó una muestra de 350 mL, que se colocó en un recipiente estéril debidamente
rotulado. Las muestras se mantuvieron en frío (4 °C a 8 °C) para su análisis inmediato (DOF, 2015c).
Análisis de indicadores bacteriológicos
Para la identificación de coliformes totales, fecales y de
las bacterias de P. aeruginosa se utilizó el
método del número más probable (NMP) con tres réplicas para P. aeruginosa y cinco réplicas para coliformes totales
y coliformes fecales (Mahapatra y col., 2015; Daley y col., 2018; Zhou y col., 2019). Para la determinación de las bacterias coliformes totales
se realizó una prueba presuntiva con caldo
Lauril sulfato (Bioxon) a 37 °C, en una incubadora (Labnet, modelo I5110A, New York, EUA), por un periodo de 24 h a 36 h. El análisis confirmatorio se llevó a cabo con caldo bilis verde brillante al 2 % (Bioxon)
a 44 °C, en una incubadora (Labnet, modelo I5110A, New York, EUA) durante 24 h (DOF,
2015c; Miyagi y col., 2017). Una muestra se consideró positiva para
coliformes totales si presentaba >
1.1 NMP para 100 mL de agua; las muestras
negativas fueron aquellas que tenían concentraciones no detectables de
bacterias o < 1.1 NMP por 100 mL de
agua (Miyagi y col., 2017).
El límite permisible de P. aeruginosa
es < 1.1 NMP por 100 mL; coliformes fecales = 0 NMP en 100 mL (Loyola y col., 2020) y para E. faecalis < 1.1 NMP en 100 mL (DOF, 2015b; Pant y col., 2016).
Se trabajó el método de goteo en placa para cuantificar
las bacterias. Se establecieron diluciones decimales seriadas para cada
muestra, con un volumen final de 1 mL. Posteriormente, se inocularon 20 mL de cada dilución en placas con medios
selectivos. Para cuantificar los coliformes totales y fecales, se utilizaron
agar MacConkey y agar eosina azul de metileno (Bioxon)
(Farkas y col., 2012; Mohamed y col., 2020). Las
bacterias P. aeruginosa se detectaron usando
un medio selectivo agar leche; y E. faecalis utilizando
agar bilis esculina (BD Difco) (DOF, 2015a; Pant y col., 2016). Las placas se mantuvieron a 35 °C, en
una incubadora (Labnet, modelo I5110A, New York, EUA)
durante 24 h.
Selección de los aislados
A partir
de las 70 muestras de agua de garrafón
colectadas y analizadas, 29 resultaron contaminadas. De los 29
garrafones con poblaciones bacteriológicas, se obtuvieron 56 cepas (en 27
garrafones se colectaron 2 cepas en cada uno, por lo que sumaron 54; y
en 2 garrafones se colectó 1 cepa por garrafón, obteniendo 2 cepas). El
criterio de selección fue la morfología colonial, se buscó que esta fuese
idéntica al interior de cada muestra.
Identificación bioquímica
Las colonias presuntivas aisladas (56) se examinaron mediante
tinción de Gram. Posteriormente, se realizó una identificación bioquímica a
través de la prueba estándar (Farkas y col., 2012) y
el kit de identificación TM Identification Systems Enteric/Nonfermenter (BBL Crystal) (Nath y
col., 2020). Los 21 aislados
identificados de acuerdo con la NOM-127-SSA1-202 como P. aeruginosa
carecieron de un perfil bioquímico preciso, 14 cepas presentaron un perfil
bioquímico similar a P. aeruginosa, en un 70 %, y 7 aislados mostraron
un perfil ambiguo (40 % de semejanza). Las 25 colonias de coliformes totales se
clasificaron, de acuerdo con su perfil bioquímico, como Enterobacter cloacae
(13) y Serratia marcenses (12), pero de estas últimas, 3 colonias
presentaron un perfil ambiguo (35 % de semejanza) para coliformes totales. A
estos 10 aislados (7 de mayor ambigüedad para P. aeruginosa y 3 para
coliformes, específcamente Serratia marcenses) se les realizó la amplificación parcial del gen 16S rRNA, para la confirmación del género bacteriano. El aislado
Escherichia sp M3 se
utilizó como control positivo para
Escherichia coli, ya
que presentó un perfil bioquímico bien
definido.
Identificación molecular del gen 16S rRNA
La identificación molecular de las muestras que fueron ambiguas
en el género bacteriano (n = 10) se realizó mediante el análisis de secuencias
parciales del gen 16S rRNA. El ADN se extrajo usando
un kit de purificación de ADN genómico (Promega Co., EE. UU.). Los oligonucleótidos
conservados empleados para la reacción de PCR fueron fD1 (5´-AGAGTTTGATCCTGGCTCAG-3´)
y rD1 (5AAGGAGGTGATCCAGCC-3´). Para ello, se utilizó el reactivo Master MIX (Invitrogen), que amplifica un fragmento de
1500 pb, que corresponde al 90 % de la
longitud del gen 16S rDNA, en un termociclador
(BioRad, T100, Berkeley, Estados Unidos de América) (Chenia y Duma, 2017). La
amplificación por PCR se llevó a cabo de la
siguiente forma: desnaturalización inicial a
94 °C durante 5 min, seguida de 30 ciclos de desnaturalización a 94 °C durante
30 s, alineamientoa
59 °C durante 30 s y extensión a 72 °C durante
1 min. Se realizó una etapa de extensión de 10 min a 72 °C después del último
ciclo, para asegurar la síntesis
completa del fragmento. La amplificación del gen se verificó con una electroforesis en gel de agarosa al 1 %,
y la tinción se realizó con bromuro de etidio
al 0.5 %. El marcador de peso molecular empleado para verificar el tamaño del amplificado fue de 1 Kb (Thermo
Scientific).
El gel fue visualizado bajo luz ultravioleta
con un fotodocumentador (Witeg,
WGD-30, Korea).
Los genes purificados se enviaron a secuenciar
a la Unidad de Síntesis y Secuenciación de DNA- UNAM.
Las secuencias parciales se analizaron con el programa
informático denominado herramienta de búsqueda de alineación local básica
(BLAST, por sus siglas en inglés:
Basic Local Alignment Search
Tool) del Centro Nacional para la Información Biotecnológica, que es parte de
la Biblioteca Nacional de Medicina de Estados Unidos (NCBI, por sus siglas en
inglés: National Center for Biotechnology Information). El programa BLAST
permitió identificar el género bacteriano de los aislados, mediante la búsqueda
de secuencias similares en la base de datos internacional del GenBanck del NCBI. Los parámetros que indicaron que los
aislados pertenecían a un determinado género fueron el porcentaje de identidad
superior al 97 % y un valor de E igual a 0 (Aziz y
col., 2017; Nath y col., 2020). Las secuencias
parciales de los 7 aislados de Pseudomonas se alinearon con las secuencias de
otros organismos de esta misma especie. Los 3 aislados incialmente
identificados como coliformes (Serratia marcenses) se
alinearon con especies de Aeromonas. Lo anterior de
acuerdo con las secuencias disponibles en la base de datos del GenBank. Las secuencias utilizadas correspondieron a los siguientes
números: MF716716.1, MK652096.1 y KROO6248.1 para Aeromonas;
MH211267.1, KJ756337.1, MH304251.1, MG833399.1 y AF390747.1 para las diferentes
especies de Pseudomonas; y para Escherichia
sp MH465145.1.
Las
secuencias parciales de los 10 aislados y las especies relacionadas
filogenéticamente fueron analizadas por alineamientos múltiples de secuencia,
para esta determinación se utilizó el programa CLUSTAL-X, y la historia
evolutiva se especificó con la construcción de un árbol filogenético y se
aplicó el algoritmo de máxima verosimilitud del programa MEGA (versión 7).
Ensayo de cristal violeta para biopelícula en microplacas
La formación de biopelícula en microplacas se
cuantificó mediante la tinción cristal violeta
reportada por O’Toole y Kolter
(Elhariry y col., 2012; Huerta y col., 2016). Los
cultivos se dejaron crecer durante una noche (18 h) y se diluyeron en una
proporción de 1:1000 en caldo glicerol fosfato (GP), las diluciones se
transfirieron a microplacas de 96 pozos, a un volumen de 200 L por pozo.
Posteriormente, la placa se mantuvo durante 48 h a 37 °C sin agitación, en una
incubadora (Labnet, modelo I5110A, New York, EUA).
Los pozos fueron aspirados y lavados con agua destilada y se secaron al aire.
Las biopelículas se tiñeron durante 15 min con 250 L de solución de cristal
violeta al 1 %. Los pozos se lavaron minuciosamente con agua corriente y
se dejaron secar al aire. La cuantificación del cristal violeta se realizó
solubilizando al cristal violeta con etanol
durante 30 min y se midió la absorbancia
a la densidad óptica de 620 nm, en un espectrofotó metro de ELISA (ThermoLab,
LabX Mutiskan, Ontario, Canada). La
formación de la biopelícula fue normalizada con la concentración de
proteína en función de la densidad óptica (DO/mg
de proteína), que fue medida con el espectrofotómetro
(DLAB, SP_UV-1100, Beijing, China). Cada determinación se realizó con 9
repeticiones. La cepa control utilizada
fue Pseudomonas putida KT2440, cepa
formadora de biopelícula, característica previamente documentada (Liu y col., 2017).
Se establecieron tres niveles de producción de biopelícula
utilizando los criterios de clasificación de Basson
y col. (2008): nivel bajo³ 0.2 y £ 0.3, nivel
moderado > 0.3 y < 0.8, nivel alto > 0.8 de DO/mg de proteína.
Prueba de
resistencia a antibióticos
Se empleó el método de difusión en disco para 12
agentes antimicrobianos (Multibac I.D): Nitrofurantoína (NF 300 mg), Sulfametoxazol (SXT 25 mg), Cloranfenicol (CL 30 mg), Norfloxacina
(NOF 10 mg),
Ciprofloxacina (CPF 5 mg), Netilmicina (NET 30g), Cefotaxima (CFX 30 mg), Cefalotina
(CF 30 mg),
Gentamicina (GE 10 mg), Carbenicilina (CB 100 mg), Amikacina (AK 30 mg) y
Ampicilina (AM 10 mg).
Las placas se mantuvieron durante 48 h a 37 °C en incubadora Labnet (modelo I5110A, New York, EUA), siguiendo
las técnicas descritas por Mohamed y col.
(2020); Nowrotek y col. (2021). La sensibilidad fue interpretada de acuerdo con los estándares del
Instituto de Normas de Laboratorio Clínico (Nowrotek
y col., 2021). La cepa control utilizada fue Pseudomonas
putida KT2440 por su multirresistencia
a antibióticos (Baltrus y col.,
2021).
Detección de hemólisis
Se analizó la presencia del factor de virulencia de
reacción hemolítica de eritrocitos. Se utilizó la base del agar sangre (Bioxon) más el 5 % de eritrocitos de
conejo. Los aislados bacterianos fueron
sembrados y se mantuvieron a 37 °C en incubadora Labnet
(modelo I5110A, New York, EUA),
durante 24 h a 48 h (Skwor y col., 2014: Nath
y col., 2020). La cepa control utilizada fue Pseudomonas putida
KT2440, carente de capacidad hemolítica, característica que se evidenció en este estudio. El tipo
de hemólisis presente en las cepas se
caracterizó como a (parcial), b (completa), o g (ausente).
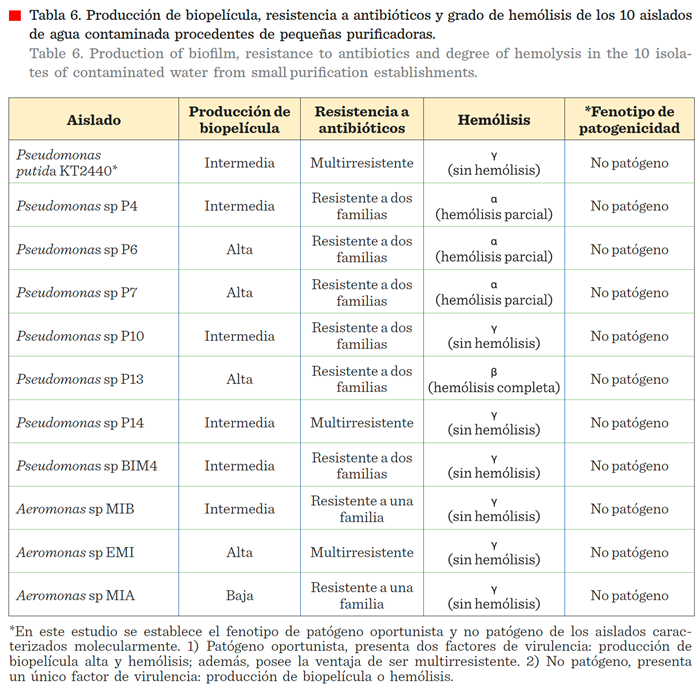
Criterios de patogenicidad
Los criterios establecidos en este experimento para designar
a la bacteria como patógeno oportunista fueron: presentar dos factores de
virulencia, 1) producir biopelícula en alta concentración y 2) mostrar
actividad hemolítica; aunado a la ventaja de la multirrestencia,
resistente al menos a un antibiótico de tres o más familias diferentes. En
contraste, una bacteria se considera no patógena cuando presenta un solo factor
de virulencia y no posee multirresitencia a
antibióticos.
Datos y análisis estadístico
La calidad bacteriológica del agua se examinó
siguiendo los métodos de las Normas Oficiales
Mexicanas y de la OMS (2017). Se calcularon la media aritmética y el
error estándar. Las mediciones de incidencia de contaminación se describieron
mediante frecuencias relativas. La incidencia de los indicadores bacteriológicos
fue analizada por región mediante una
comparación de Chi-cuadrado, teniendo como variables categóricas a las
filas (x): las zonas NP y SO y, columnas (y): el tipo de indicador microbiológico, agrupados en 3 categorías: 1) coliformes totales, 2) coliformes fecales más Enterococus
faecalis y 3) Pseudomonas
sp). La biopelícula producida por las
10 cepas bacterianas y el grupo control, fue comparado mediante un análisis de varianza (ANOVA) paramétrico
de una vía. Los datos fueron transformados
con la ecuación: log (x)*2.403 6, para
cumplir con los supuestos de
normalidad y homocedasticidad. Se hicieron las comparaciones a posteriori a través de la prueba de diferencias honestamente significativas
(HSD, por sus siglas en inglés: Honestly Significant Difference) de Tukey-Kramer (Software JMP 10.0.0, Copyright© 2012 SAS Institute Inc.).
RESULTADOS
Indicadores bacteriológicos
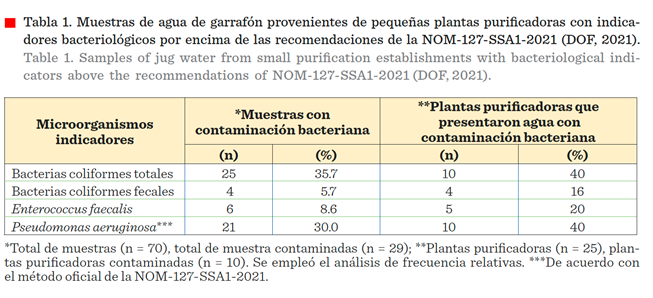
El
análisis bacteriológico de las 70 muestras de agua indicó que 29 de ellas (41.4
%) estuvieron contaminadas y no cumplieron
con la NOM-127-SSA1-2021 (DOF, 2021), así como las normas
internacionales (Tabla 1), en tanto que 41 (58.6 %) de ellas si cumplieron con
la calidad microbiológica adecuada. De las 70
muestras (Tablas 1 y 2), únicamente 25
(35.7 %) presentaron contaminación por coliformes totales y 4 (5.7 %)
reportaron bacterias coliformes fecales, mientras que 21 (30 %) registraron Pseudomonas aeruginosa
y 6 (8.6 %) E. faecalis. Los garrafones
de agua contaminados con bacterias coliformes totales y Pseudomonas se
reportaron en 40 % (n = 10) de las 25 plantas purificadoras evaluadas; 4 de estas
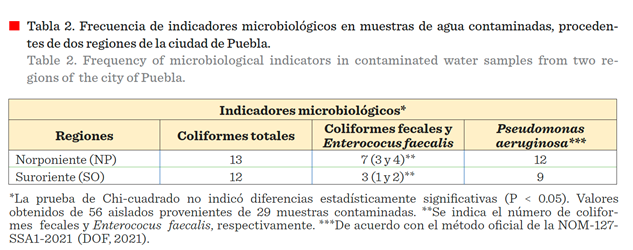
10 plantas presentaron bacterias coliformes fecales y 5 E. faecalis (Tabla 1). El análisis de Chi-cuadrado indicó
que no hubo diferencia significativa (P < 0.05) entre el tipo de indicador
microbiológico detectado para cada zona de la ciudad (NP y SO)
(Tabla 2).
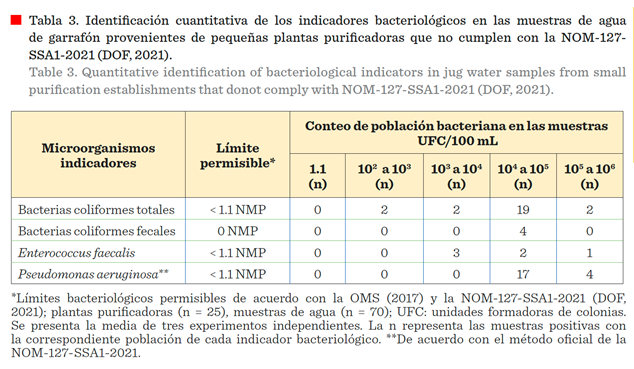
Las
muestras de agua presentaron una población
bacteriana de coliformes totales entre 102 NMP/100 mL a 106
NMP/100 mL, mientras que para coliformes
fecales la población bacteriana fluctuó entre 104 NMP/100 mL a 105
NMP/100 mL. Las muestras con presencia de E. faecalis
registraron poblaciones entre 103
NMP/100 mL a 106 NMP/100 mL;
mientras que las poblaciones de P.
aeruginosa se encontraron en el orden de 104 NMP/100 mL
a 106 NMP/100 mL (Tabla 3).
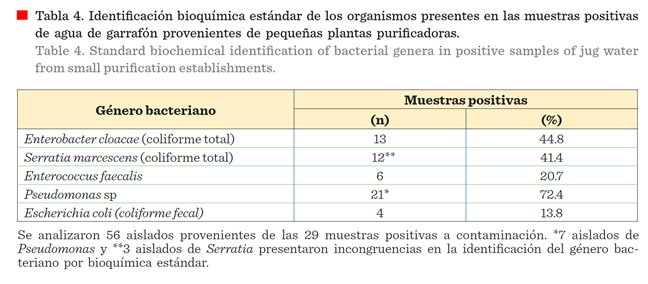
Identificación bioquímica
En el caso P. aeruginosa,
se reportaron 21 organismos presentes en las muestras
contaminadas mediante la
técnica indicada por la NOM-201-SSA1-2015 (DOF,
2015b) (Tabla 4). La norma establece
como prueba positiva para esta especie
la producción de sideróforos en el medio selectivo
agar leche, visualizados como pigmentos fluorescentes amarillo-verdoso.
Sin embargo, al realizar la identificación
bioquímica estándar para dicha bacteria, se detectaron discrepancias en las pruebas enzimáticas para la degradación
de la fuente de carbono y de aminoácidos, así como, la producción de
otros sideróforos, que dificultaron su confirmación. Se encontraron 7 cepas que mostraron elevada inconsistencia en las pruebas bioquímicas confirmatorias
para Pseudomonas aeruginosa.
Los 56
aislados obtenidos de las 29 muestras de agua fueron identificados mediante
pruebas microbiológicas y reportados en la
Tabla 4. Los organismos E. cloacae, E.
faecalis y E. coli fueron corroboradas bioquímicamente. En el caso de
S. marcescens (coliformes totales), 3 cepas no
pudieron ser confirmadas, ya que mostraron
variaciones en la degradación de la fuente de carbono y aminoácidos.
Análisis molecular
El
análisis molecular identificó que los aislados inicialmente identificados como P.
aeruginosa, mediante la NOM-201-SSA1-2015, si pertenecen al género Pseudomonas, pero se
sugiere que están relacionados filogenéticamente con las especies P. alcaligenes, P. salomonii, P. fluorescens y
P. poae (Figura 1). Por otra parte, los aislados
identificados inicialmente como S. marcescens
(aislados EMI, MIA y MIB) en realidad forman parte del género Aeromonas y presentan relación filogenética con las
especies Aeromonas veronii,
Aeromonas caviae y
Aeromonas hydrophila.

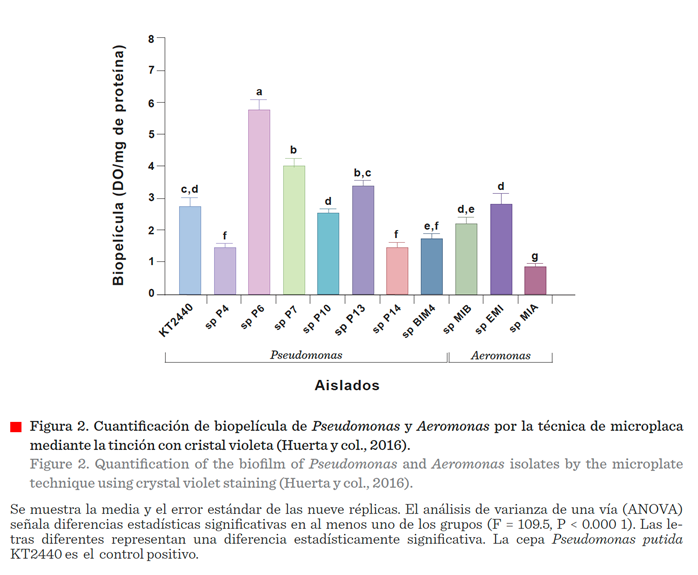
Cuantificación de biopelícula
La
presencia y la cuantificación de la biopelícula
se le determinó a los 10 aislados confirmados por estudios moleculares y a la cepa control Pseudomonas putida
KT2440; todos los aislados presentaron producción de biopelícula. En la
comparación de la producción de biopelícula
entre los aislados se detectó diferencia entre los aislados (F 0.05,
10,87 = 109.5, P < 0.000 1). La comparación a posteriori de Tukey-Kramer indicó que el aislado Pseudomonas
sp P6 presentó significativamente los niveles más elevados de producción
de biopelícula con un promedio de 5.87
(DO)/mg de proteína, y el aislado de Aeromonas
sp MIA presentó los
niveles significativamente más bajos
(P < 0.001) con 0.90 (DO)/mg de proteína (Figura 2).
Resistencia a antibióticos y hemólisis
Las cepas
con resistencia a un mayor número de antibióticos fueron el control P. putida KT2440 y el aislado Pseudomonas sp P14,
que son resistentes a 9 y 8 antibióticos, respectivamente (Tablas 5 y 6). Los
aislados del género Pseudomonas son resistentes a norfloxacina,
excepto la cepa control KT2440 y el aislado de Aeromonas
sp MIA.
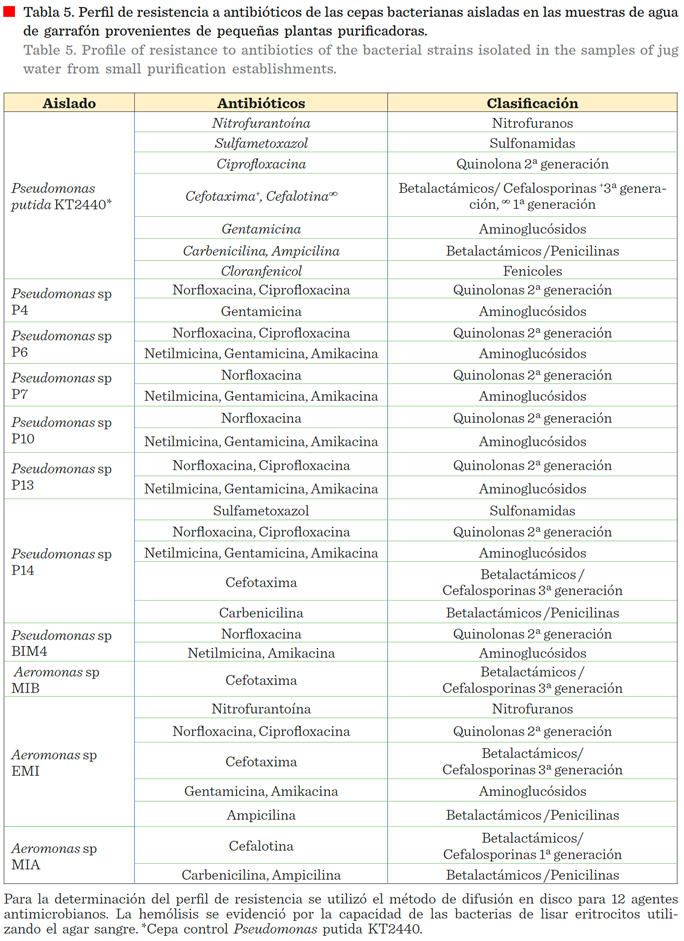
Los aislados de Pseudomonas sp
P14 y Aeromonas sp
EMI son bacterias multirresistentes, así como la cepa
control P. putida KT2440 (Tabla 6), en contraste, los aislados Pseudomonas
sp BIM4 y Pseudomonas sp P4 presentaron la mayor susceptibilidad
a antibióticos (Tabla 5). La tendencia de la
mayoría de los aislados de Pseudomonas sp mostraron resistencia a
dos familias de antibióticos, y los tres aislados de Aeromonas
MIB y MIA fueron
resistentes a betalactámicos.
Los
aislados del género Pseudomonas sp P4, P6
y P7 presentaron hemólisis parcial. La cepa KT2440 (control), P10, P14 y
BIM4 no presentaron hemólisis y solo el
aislado de Pseudomonas sp
P13 presentó hemólisis completa. En el caso
del género Aeromonas, ninguno aislado presentó
hemólisis (Tabla 6).
En este estudio, los 10 aislados caracterizados
molecularmente fueron clasificados como no
patógenos, dado que se requieren al menos 2 factores de virulencia y la multirresistencia a antibióticos para considerarlos como patógenos
oportunistas (Tabla 6).
DISCUSIÓN
El 40 %
de las pequeñas plantas purificadoras de la ciudad de Puebla, México,
comercializaron agua contaminada con
coliformes fecales, coliformes totales, E. faecalis
y Pseudomonas sp (Tablas 1 y 2), de acuerdo
con la NOM-127-SSA1-2021. Existen pocos
estudios que analizan la presencia de
indicadores bacteriológicos en el agua de garrafón proveniente de pequeñas plantas purificadoras. Abada y col.
(2019); Cerna-Cortes y col. (2019), determinaron
la calidad bacteriológica del agua
obtenida de pequeños establecimientos dedicados a su purificación, de Arabia Saudita y de la Ciudad de
México, respectivamente, y encontraron bacterias coliformes totales en más del
60 % de sus muestras, y entre el 3 % y el 23 % de las muestras presentaron coliformes fecales, los cuales concuerdan con
lo aquí reportado.
En el presente estudio, se registraron seis muestras de
agua con contaminación por E. faecalis.
Algunas investigaciones demostraron que esta
bacteria se caracteriza por su capacidad de
transferir genes que codifican para la resistencia a antimicrobianos (Chacón y
col., 2018; Cho y col., 2020), a pesar de que no cuenta con un potencial alto de virulencia (Enayati
y col., 2015).
Existen reportes que establecen la presencia de Enterococcus sp en muestras de agua del grifo de Dharan, Nepal (Pant y col.,
2016); por lo anterior, el monitoreo de esta bacteria es importante, ya que
corrobora la presencia de contaminación fecal.
El
análisis de Chi-cuadrado indicó la falta de asociación entre las zonas NP y SO
con el indicador bacteriológico, mostrando similitud en la presencia de las
bacterias contaminantes en ambas zonas. En el estudio de Tyagi
y col. (2015) se realizó la comparación de coliformes fecales y totales en dos
regiones del estado de Uttarakhand en la India, Garhwal y Kumaun; encontrando un
mayor grado de contaminación en la región Kumaun. No
obstante que la investigación de Tyagi se llevó a cabo en agua proveniente de cuerpos de agua naturales.
Otros trabajos comparan los marcadores microbiológicos
en diferentes fuentes, como tinacos municipales y botellas de agua, sin
considerar el origen por zonas (Pant y col., 2016). Las investigaciones que contrastan el agua
proveniente de agua embotellada por regiones
son escasas en la literatura y este estudio contribuye
a establecer la existencia de indicadores
microbiológicos por zonas.
Las muestras contaminadas reportaron distintas
especies de Pseudomonas diferentes a P. aeruginosa, además de Aeromonas
(Figura 1). Asimismo, solo 2 organismos aislados, más el control,
mostraron resistencia a más de dos familias de antibióticos, por lo que se conside-ran bacterias multirresistentes; además produjeron
biopelícula.
Los
resultados indicaron que las muestras de agua contaminadas presentaron hasta
100 000 veces más bacterias coliformes
totales (Tabla 3) que lo estipulado en las normas nacionales e internacionales.
Los coliformes totales (Klebsiella, Citrobacter, Enterobacter, Serratia y Proteus) son indicadores
bacteriológicos que están estrechamente relacionados con la contaminación ambiental de suelos, ríos, superficies foliares
de plantas y aguas residuales (Wu y col., 2011). Además, un porcentaje pequeño
de las muestras de agua contaminada con
bacterias coliformes totales (5.7 %), reportó altas poblaciones de E.
coli (Tabla 3). Dicha
bacteria es representativa de las coliformes fecales (DOF, 2015b), lo
que sugiere la existencia de contaminación fecal en el agua (Daley y col.,
2018). La presencia de E. coli está
estrechamente relacionada con el aumento de infecciones gastrointestinales en
la población (Saxena
y col., 2015) por lo que no debería estar en el agua para consumo humano
(Chacón y col., 2018).
Dentro de los indicadores bacteriológicos considerados
estratégicos al determinar la calidad de agua para consumo humano, está el
análisis de
P. aeruginosa. El género Pseudomonas es
ubicuo en la naturaleza y se encuentra comúnmente en la microflora autóctona de las aguas, incluso en
aquellas con bajos niveles de nutrientes (Camiade y
col., 2020; Nordstedt y col., 2020).
Los
resultados de este estudio señalaron que 21 de las muestras de agua embotellada
contaminadas con coliformes totales presentaron poblaciones de Pseudomonas
sp, que superaron en 100 000 veces los límites permisibles establecidos por las normas oficiales. Además,
los resultados sugieren que el agua embotellada puede ser un vector de
transmisión del género Pseudomonas que podrían causar enfermedades en
grupos vulnerables y la descomposición de alimentos (Wu y
col., 2011).
La
versatilidad metabólica que presentan el género Pseudomonas y el grupo
de las Entero-bacterias, en algunas ocasiones, hace
variar la producción de metabolitos secundarios; por lo tanto, el perfil
bioquímico de estas especies no siempre coincide con lo establecido en la literatura
(Sudan y col., 2018); por ello, se realizaron
análisis moleculares para corroborar la identidad del género de los microorganismos aislados.
El análisis filogenético de los 7 aislados de Pseudomonas
mostró cercanía con las especies P. alcaligenes,
P. salomonii, P. fluorescens
y P. poae. Al respecto, Woodring
y Farrell (2019) reportaron que P. fluorescens
y P. poae tienen el potencial genético de ser
patógenos oportunistas. Por otra parte, Sala-Comomera
y col. (2016), aislaron a otras especies del género Pseudomonas, sin aislar a P. aeruginosa,
y concluyeron que la comunidad bacteriana identificada en el agua purificada por ósmosis reversa no representa
un riesgo para la salud (Sacchetti y col., 2015). Es
importante, por lo tanto, determinar si las especies de Pseudomonas,
aisladas en el presente estudio, presentaban patogenicidad. Otro microorganismo
de interés para la salud pública es Aeromonas
sp, que se aísla de forma ubicua de muestras de carne,
leche, productos lácteos y agua (Vávrová y col., 2015; Stratev
y Odeyemi, 2016). En el presente trabajo se detectó la existencia
de cepas de 3 Aeromonas, por lo que se
consideró el determinar si mostraban
patogenicidad.
En este
estudio se identificaron aislados muy cercanamente relacionados con las
especies: A. hydrophila, A. caviae
y A. veronii, que estaban presentes en las
muestras contaminadas con coliformes totales. Las características bioquímicas
de las especies del género Aeromonas sp son similares, lo que dificulta la caracterización y diferenciación fenotípica (Vávrová y
col., 2015). Aeromonas hydrophila
y A. caviae son
patógenos acuáticos oportunistas que causan infecciones gastrointestinales
(Horn y col., 2016)
y septicemia en humanos y animales (Stratev y Odeyemi, 2016; Zhou y col., 2019). Los estudios que
analizan el género Aeromonas en agua embotellada
para consumo humano son prácticamente inexistentes. Sin embargo, este género se
ha identificado en el agua potable y tiene la capacidad de producir enterotoxinas (Farkas y col.,
2012; Miyagi y col., 2017) y su presencia se ha asociado con diarrea infantil
(Aziz y col., 2017).
La
Agencia de Protección Ambiental de los Estados Unidos, ha colocado al género Aeromonas en la lista de especies candidatas a contaminantes,
por lo que la OMS ha propuesto el uso de esta bacteria como marcador de riesgo
en los sistemas de distribución de agua (OMS, 2017). Cabe mencionar, que ni el
género Aeromonas ni otras especies distintas a
P. aeruginosa, están reportadas en las normas
oficiales como indicadores bacteriológicos
del agua para consumo humano; no obstante,
han sido catalogadas como bacterias oportunistas que pueden ocasionar daños
en pacientes inmunocomprometidos (Wu y col., 2011; Govender
y col., 2021).
En este
estudio, se analizó la producción de biopelícula
en los 4 géneros Pseudomonas y 3 de Aeromonas;
ambas especies en general mostraron niveles altos e intermedios (Figura
2, Tabla 6). La biopelícula favorece la colonización de ambos géneros a su
hábitat, independientemente de si se
trata de cepas patógenas o no patógenas (Maes y col.,
2020) y, constituye un ambiente idóneo para la transferencia horizontal de
genes de resistencia a antibióticos (Mulamattathil y col., 2014). La cantidad de biopelícula
depende de varios factores, y los
más reportados son la temperatura, aireación, disponibilidad de nutrientes y la
especie; por ejemplo, Chenia y Duma (2017) observaron
que en los aislados de Aeromonas procedentes de pescados y agua de mar, la cantidad
de biopelícula producida a 30 °C depende de la especie. De forma similar, Elhariry y col. (2012)
aislaron a Pseudomonas a partir de agua potable, la cual produjo biopelícula de forma moderada a 30 °C. Esta tendencia también se observó en el presente estudio, con
los aislados de Aeromonas y Pseudomonas,
lo que corrobora que la cantidad de biopelícula depende de
la especie.
Pseudomonas se
caracteriza por ser multirresistente a los antibióticos (Horn
y col., 2016; Camiade y col., 2020); sin embargo, los
resultados de este estudio muestran que la mayoría de los aislados de Pseudomonas
son resistentes únicamente a dos grupos de antibióticos: Quinolonas (norfloxacina y ciprofloxacina)
y Aminoglucósidos (gentamicina, amikacina y netilmicina), por lo que no se podrían considerar como
bacterias multirresistentes. Solo dos excepciones mostraron multirresistencia
a antibióticos: un aislado de Pseudomonas (P14) y un aislado de Aeromonas
(EMI) (Tabla 6). El trabajo de Sala-Comorera y col. (2016) señaló que el 95 % de los aislados en agua purificada
fueron resistentes a cloranfenicol, aztreonam y cefotaxima.
Camiade y col. (2020) reportaron que los aislados provenientes de restos fecales de humanos y
ganado manifestaron resistencia a betalactámicos (penicilinas, cefalosporinas y
monobactámicos), fenicoles (cloranfenicol) y sulfonamidas
(trimetroprima y sulfametoxazol), pero fueron sensibles
a quinolonas y aminoglucósidos, contrario a
lo que se reporta en esta investigación. La tendencia observada en la
Tabla 5 es semejante al patrón de resistencia a
cefalosporinas, cefixima, trimetroprima
y sulfametoxazol, en aislados de aguas superficiales y residuales
reportado por Govender y col.(2021).
El
aislado de Aeromonas (EMI) presentó multirresistencia a los antibióticos (gentamicina, ciprofloxacina
y betalactámicos), semejante al perfil de
resistencia de los aislados reportados en heces fecales y alimentos por Kamal y Abdel-Latef (2015) y en el 51.6 % de los aislados provenientes
de agua de ambientes acuícolas obtenidos por Dhanapala
y col. (2021). La resistencia a penicilina y ampicilina se ha reportado en el 100 % de los aislados de Aeromonas a partir de granjas piscícolas (Dhanapala y col., 2021), al igual que en la presente
investigación.
La
actividad hemolítica fue detectada en más de la mitad de los 7 aislados de Pseudomonas,
en este estudio. La hemólisis en Pseudomonas tiende a presentarse
con frecuencia en cepas que se comportan como patógenos oportunistas; por ejemplo, en P. aeruginosa
aislada a partir de basura proveniente de hospital (Nath
y col., 2020), en aislados clínicos de P. aeruginosa y P. fluorescens corroborados en
cultivo celular (Rossignol y col., 2008). Un
ejemplo particular, es P. fluorescens, carece
de hemólisis cuando se comporta como saprófita, contrario a las cepas
oportunistas provenientes de aislados clínicos que sí presentan hemólisis (Sperandio y col., 2012). Los aislados de Aeromonas en este trabajo no generaron hemólisis, a
diferencia de lo reportado en los aislados de A. caviae
a partir de aguas residuales, que producen
beta-hemólisis, biopelícula y multirresistencia (Nowrotek y
col., 2021). La actividad hemolítica de Aeromonas
ha sido comprobada en diversos estudios como en pescados y agua de mar (Kamal y Abdel-Latef, 2015), en
alimentos y muestras de heces fecales de humanos (Chenia y
Duma, 2017).
De
acuerdo con la evidencia en la literatura, para
asignar a una bacteria como patógena oportunista se requiere evidenciar
al menos tres factores de virulencia (Rossignol y
col., 2008; Nowrotek y col., 2021). En este estudio se
consideraron como patógenos oportunistas a las bacterias aisladas que
presentaron los factores de virulencia, biopelícula y hemólisis, y la multirresistencia a
antibióticos (Tabla 6). Con estas características ningún aislado fue clasificado como patógeno oportunista. Sin embargo, destaca la presencia de la Pseudomonas sp
P 13, con alta producción de biopelícula y hemólisis completa, la cual
al ser resistente a dos familias de
antibiótico podría afectar a pacientes inmunocomprometidos, si el tratamiento
adecuado no se da de manera oportuna.
CONCLUSIONES
El 40 %
de las pequeñas plantas purificadoras de agua no cumplieron con la normativa de
la calidad de agua para consumo humano.La
presencia de Aeromonas y Pseudomonas fue
identificada en las muestras contaminadas. Los aislados de estos géneros
bacterianos no se consideraron patógenos oportunistas al no presentar las tres características evaluadas, sin
embargo, destaca el amplio número de
antibióticos a los cuales se están volviendo resistentes. Es recomendable
ampliar la caracterización a nivel molecular y probar la patogenicidad de los
aislados de Aeromonas y Pseudomonas en un
modelo animal.
AGRADECIMIENTOS
Se
agradece a la Facultad de Ciencias Biológicas, Benemérita Universidad Autónoma
de Puebla, por el apoyo parcial vía
distribución del presupuesto designado a la investigación.
Referencias
Abada,
E., Al-Fifi, Z., Al-Rajab,
A. J., Mahdhi, M., and Sharma, M.
(2019). Molecular identification of biological
contaminants in different drinking water resources of the Jazan
region, Saudi Arabia. Journal of Water and Health. 17(4): 622-632.
Aziz, F., Parrado, R. J., Ouazzani, N., Dary, M., Manyani, H., Rodriguez,
M. B., and Mandi, L. (2017). Sanitary impact evaluation of drinking water in
storage reservoirs in Moroccan rural area. Saudi Journal of Biological
Sciences. 24(4): 767-777.
Baltrus, D. A., Smith, C., Derrick, M., Leligdon, C.,
Rosenthal, Z., Mollico, M…., and Clark, M. (2021). Genomic
Background Governs Opposing Responses to Nalidixic Acid upon Megaplasmid Acquisition in Pseudomonas. MSphere.
6(1): e00008-21.
Basson, A., Flemming, L. A.,
and Chenia, H. Y. (2008).
Evaluation of Adherence, Hydrophobicity, Aggregation, and Biofilm Development
of Flavobacterium johnsoniae-Like Isolates. Microbial Ecology. 55(1):
1-14.
Camiade, M., Bodilis, J., Chaftar,
N., Riah-Anglet, W., Gardères,
J., Buquet, S., …, and Pawlak, B. (2020). Antibiotic
resistance patterns of Pseudomonas spp. isolated
from faecal wastes in the environment and
contaminated surface water. FEMS Microbiology Ecology. 96(2):
fiaa008.
Cerna-Cortes,
J. F., Cortes-Cueto, A. L., Villegas-Martínez,
D., León-Montes, N., Salas-Rangel, L. P., Rivera-Gutiérrez, S., …, and
González, Y. M. J. A. (2019). Bacteriological quality of bottled
water obtained from Mexico City small water purification plants: Incidence
and identification of potentially pathogenic
nontuberculous mycobacteria species. International Journal of Food
Microbiology. 306: 108260.
Chacón, L., Arias, V., Barrantes, K., Beita-Sandí, W., Reyes, L., and Achí,
R. (2018). Enterococci as a key parameter for
water quality index: Purires River, Costa
Rica. Journal of Water and Health. 16(6): 1007-1017.
Chenia,
H. Y. and Duma, S. (2017). Characterization of virulence, cell surface
characteristics and biofilm-forming ability of Aeromonas spp. Isolates from
fish and sea water. Journal of Fish Diseases. 40(3): 339-350.
Cho, S., Jackson, C. R., and Frye, J. G. (2020).
The prevalence and antimicrobial resistance phenotypes of
Salmonella, Escherichia coli and Enterococcus sp. in surface water. Letters
in Applied Microbiology. 71(1): 3-25.
Daley, K., Truelstrup-Hansen, L., Jamieson, R.
C., Hayward, J. L., Piorkowski, G. S., Krkosek, W., …, and Huang Y. (2018). Chemical and microbial
characteristics of municipal drinking water supply systems in the Canadian
Arctic. Environmental Science and Pollution Research. 25(33): 32926-32937.
Dhanapala, P. M., Kalupahana, R. S., Kalupahana, A. W., Wijesekera, D.
P. H., Kottawatta, S. A., Jayasekera, N. K., ..., and
Jagoda, S. D. S. (2021). Characterization and Antimicrobial
Resistance of Environmental and Clinical Aeromonas Species Isolated from Fresh
Water Ornamental Fish and Associated Farming
Environment in Sri Lanka. Microorganisms.
9(10):
2106.
DOF,
Diario Oficial de la Federación (2015a). NOM-210-SSA1-2014, Productos y
servicios. Métodos de prueba microbiológicos. Determinación de microorganismos
indicadores. Determinación de microorganismos patógenos, en Diario Oficial
de la Federación. [En línea]. Disponible en: https://dof.gob.mx/nota_detalle.php?codigo=5398468&fecha=26/06/2015#gsc.tab=0. Fecha de consulta: 6 de abril de
2021.
DOF,
Diario Oficial de la Federación (2015b). NOM-201-SSA1-2015, Productos y
servicios. Agua y hielo para consumo humano, envasados y a granel. Especificaciones
sanitarias, en Diario Oficial de la Federación. [En línea]. Disponible
en: https://dof.gob.mx/nota_detalle.php?codigo=5420977&fecha=22/12/2015#gsc.tab=0. Fecha de consulta: 6 de abril de
2021.
DOF,
Diario Oficial de la Federación (2015c). NMX-AA-042-SCFI-2015. Análisis de agua
- Enumeración de organismos coliformes totales, organismos coliformes fecales (Termotolerantes) y Escherichia
coli - Método del número más probable en tubos
múltiples, en Diario Oficial de la Federación. [En línea]. Disponible
en: https://www.dof.gob.mx/nota_detalle.php?codigo=5433394&fecha=18/04/2016#gsc.tab=0.
Fecha de consulta: 6 de abril de 2021.
DOF,
Diario Oficial de la Federación (2021). NOM-127-SSA1-2021.
Agua para uso y consumo humano.
Límites permisibles de la calidad del agua, en Diario Oficial de la
Federación. [En línea]. Disponible en: https://dof.gob.mx/nota_detalle.php?co-digo=5650705&fecha=02/05/2022#gsc.tab=0.
Fecha de consulta: 6 de abril de 2021.
Elhariry, H., Gherbawy,
H., El-Deeb, B., and Altalhi, A. (2012).
Molecular Identification and Biofilm-Forming Ability
of Culturable Aquatic Bacteria in Microbial Biofilms Formed in Drinking Water
Distribution Networks. Geomicrobiology Journal. 29(6): 561-569.
Enayati, M., Sadeghi, J., Nahaei, M. R., Aghazadeh, M.,
Pourshafie, M. R., and Talebi,
M. (2015). Virulence and antimicrobial resistance of Enterococcus faecium
isolated from water samples. Letters in Applied Microbiology. 61(4): 339-45.
Farkas, A., Dragan-Bularda,
M., Ciataras, D., Bocos, B.,
and Tigan, S. (2012). Opportunistic pathogens and faecal
indicators in drinking water associated biofilms in Cluj, Romania. Journal
of Water and Health.10(3): 471-483.
Govender, R., Amoah, I. D., Adegoke, A. A., Singh, G., Kumari, S., Swalaha, F. M., ..., and Stenström,
T. A. (2021). Identification, antibiotic resistance, and virulence profiling of
Aeromonas and Pseudomonas species from wastewater and surface water. Environmental
monitoring and assessment. 193(5): 294.
Gutiérrez-Del-Río, I., Marín, L., Fernández, J., Álvarez-San-Millán, M., Ferrero, F. J., Valledor, M., …, and Lombo, F. (2018). Development of a biosensor protein bullet
as a fluorescent method for fastdetection of
Escherichia coli in drinking water. PloS
One. 13(1): e0184277.
Horn, S., Pieters, R., and Bezuidenhout, C. (2016). Pathogenic features of heterotrophic plate count
bacteria from drinking-water boreholes. Journal of Water and
Health. 14(6): 890-900.
Huerta, J. M., Aguilar, I., López-Pliego, L.,
Fuentes-Ramírez, L. E., and Castañeda, M. (2016). The
Role of the ncRNA RgsA
in the Oxidative Stress Response and Biofilm Formation in Azotobacter vinelandii. Current Microbiology. 72(6): 671-679.
INEGI, Instituo Nacional de
Estadística y Geografía (2010). Compendio de información geográfica municipal
2010 de los Estados Unidos Mexicanos, Puebla, Puebla. [En línea]. Disponible
en: https://www.inegi.org.mx/app/areasgeograficas/?ag=21114#collapse-Resumen.
Fecha de consulta: 19 de octubre de 2022.
Ji, L., Li, Y., Zhang, G., and Bi, Y. (2021). Anthropogenic Disturbances Have Contributed to
Degradation of River Water Quality in Arid Areas. Water. 13(22): 3305.
Jurado-Martín, I., Sainz-Mejías, M., and
McClean, S. (2021). Pseudomonas aeruginosa: An Audacious Pathogen with an
Adaptable Arsenal of Virulence Factors. International Journal of Molecular
Sciences. 22(6): 3128.
Kamal, G. and Abdel-Latef, E. (2015). Detection
of Aerolysin, Hemolysin genes and Antimicrobial susceptibility
of Aeromonas hydrophila isolated from retial foods and human stool. Global Veterinaria
14(4): 528-534.
Liu, H., Xiao, Y., Nie, H., Huang, Q., and Chen,
W. (2017). Influence of (p) ppGpp on biofilm regulation
in Pseudomonas putida KT2440. Microbiological Research. 204: 1-8.
Loyola, S., Sanchez, J. F., Maguiña, E.,
Canal, E., Castillo, R., Bernal, M., …, and Rocha, C. A. (2020). Fecal
contamination of drinking water was associated with diarrheal pathogen carriage
among children younger than 5 years in three Peruvian rural communities. The American Journal of Tropical
Medicine and Hygiene. 102(6): 1279-1285.
Madigan, T. M., Martinko, M. J., Bender, K. S.,
Buckley, D. H., and Stahl, D. A. (2015). Brock biology of microorganism.
(Fourteenth edition). United Stateof America: Pearson
Education. 1041 Pp.
Maes,
S., De-Reu, K., Van-Weyenberg, S., Lories, B., Heyndrickx, M., and Steenackers, H. (2020). Pseudomonas
putida as a potential biocontrol agent against Salmonella Java biofilm
formation in the drinking water system of
broiler houses. BMC Microbiology. 20(1): 373.
Mahapatra, A., Padhi, N., Mahapatra, D.,
Bhatt, M., Sahoo, D., Jena, S., …, and Chayani, N.
(2015). Study of biofilm in bacteria from water pipelines. Journal of
Clinical and Diagnostic Research. 9(3): DC09-DC11.
Miyagi, K., Sano, K., and Hirai, I. (2017). Sanitary evaluation of
domestic water supply with storage tanks and detection of Aeromonas, enteric
and bacteria in domestic water facilites in Okinawa Prefecture
on Japan. Water Research. 119: 171-177.
Mohamed, A., Nyerere, A., Sang, W. K., and Ngayo,
M. (2020). Bottled water brands are contaminated with multidrug resistant
bacteria in Nairobi, Kenya. F1000 Research. 9: 1337.
Mulamattathil, S. G., Bezuidenhout, C., and Mbewe, M. (2014). Biofilm formation in surface and drinking water distribution
systems in Mafikeng, South Africa. South African Journal of Science.
110(11-12): 01-09.
Nath, S., Sinha, A, Singha, Y. S., Dey, A.,
Bhattacharjee, N., and Deb, B. (2020). Prevalence
of antibiotic-resistant, toxic metal-tolerant and biofilm-forming bacteria in
hospital surroundings. Environmental Analysis, Health
and Toxicology. 35(3): e2020018.
Nordstedt, N. P., Chapin, L. J., Taylor, C. G., and Jones, M. L. (2020).
Identification of Pseudomonas spp. That Increase Ornamental Crop Quality During
Abiotic Stress. Frontiers in Plant Science. 10: 1754.
Nowrotek, M., Jałowiecki, Ł., and Płaza,
G. (2021). Fluoroquinolone Resistance and Virulence Properties Among Wastewater Aeromonas caviae Isolates. Microbial Drug Resistance. 27(2): 179-189.
OMS,
Organización Mundial de la Salud (2017). Guías para la calidad del agua de
consumo humano: Cuarta edición que incorpora la primera adenda en Organización Mundial de la Salud. [En línea]. Disponi-ble
en: https://www.who.int/es/publications/i/item/9789241549950. Fecha de
consulta: 30 de abril de 2021.
OMS,
Organización Mundial de la Salud (2022). Agua para consumo humano, en Organización
Mundial de la Salud. [En línea]. Disponible en:
https://www.who.int/es/news-room/fact-sheets/detail/drinking-water. Fecha de
consulta: 30 de abril de 2021.
Pant,
N. D., Poudyal, N., and Bhattacharya, S. K. (2016). Bacteriological
quality of bottled drinking water versus municipal tap water in Dharan municipality,
Nepal. Journal of Health, Population and Nutrition. 35(1): 1-6.
Pichel,
N., Vivar, M., and Fuentes, M. (2019). The problem of
drinking water access: A review of disinfection technologies with an emphasis
on solar treatment methods. Chemosphere. 218: 1014-1030.
Rossignol, G., Merieau,
A., Guerillon, J., Veron,
W., Lesouhaitier, O., Feuilloley,
M., and Orange, N. (2008). Involvement of a phospholipase C in the hemolytic activity of a clinical strain of Pseudomonas
fluorescens. BMC Microbiology. 8(1): 189.
Sacchetti, R., De-Luca, G., Guberti, E., and Zanetti,
F. (2015). Quality of Drinking Water Treated at Point of Use in Residential
Healthcare Facilities for the Elderly. International Journal of Environmental Research and Public Health. 12(9): 11163-11177.
Sala-Comorera, L.,
Blanch, A. R., Vilaró, C., Galofré,
B., and García-Aljaro, C.
(2016). Pseudomonas-related populations associated with reverse osmosis in
drinking water treatment. Journal of Environmental Management. 182:
335-341.
Saxena, G., Bharagava, R. N., Kaithwas, G., and Raj, A. (2015). Microbial indicators, pathogens and methods for their monitoring in water
environment. Journal of Water and Health. 13(2): 319-339.
Skwor,
T., Shinko, J., Augustyniak, A., Gee, C., and Andraso, G. (2014). Aeromonas hydrophila
and Aeromonas veronii predominate among potentially
pathogenic ciprofloxacin and
tetracycline resistant aeromonas isolates from Lake Erie. Applied and Environmental
Microbiology. 80(3): 841-848.
Sperandio, D., Decoin, V., Latour, X., Mijouin, L., Hillion, M., Feuilloley, M. G., ..., and Merieau,
A. (2012).
Virulence
of the Pseudomonas fluorescens clinical strain MFN1032 towards Dictyostelium discoideum and
macrophages in relation with type III secretion system. BMC Microbiology.
12(1): 1-10.
Stratev, D. and Odeyemi, O. A. (2016). Antimicrobial
resistance of Aeromonas hydrophila isolated from
different food sources: A mini-review. Journal of
Infection and Public
Health. 9(5): 535-544.
Sudan, S. K., Pal, D., Bisht, B., Kumar, N., Chau-dhry,
V., Patil, P., ..., and Krishnamurthi, S. (2018). Pseudomonas fluvialis
sp. nov., a novel member of the genus Pseudomonas
isolated from the river Ganges, India. International Journal of Systematic and
Evolutionary Microbiology. 68(1): 402-408.
Tyagi, S., Singh, P., Sharma, B., Singh, R., Dobhal, R., and Uniyal, D. P.
(2015). Bacteriological Assessment of Drinking Water
Sources of Uttarakhand, India. National
Academy Science Letters-India.
38(1): 37-44.
Vávrová, A., Balážová, T., Sedláček,
I., Tvrzová, L., and Šedo,
O. (2015). Evaluation of the MALDI-TOF MS profiling for identification of newly
described Aeromonas spp. Folia Microbiologica.
60(5): 375-383.
Woodring, T. S. and Farrell, J. J. (2019). Pseudomonas poae–Associated Fatal Septic Transfusion Reaction, Peoria,
Illinois, USA, 2017. Emerging Infectious Diseases. 25(8): 1445-1451.
Wu, J., Long, S. C., Das, D., and Dorner, S. M. (2011). Are microbial
indicators and pathogens correlated? A statistical analysis of 40 years of
research. Journal of Water and Health. 9(2): 265-278.
Zhou, H., Gai, C., Ye, G., An, J., Liu, K., Xu,
L., and Cao, H. (2019). Aeromonas hydrophila, an Emerging Causative Agent of Freshwater-Farmed
white leg shrimp Litopenaeus vannamei. Microorganisms. 7(10): 450.